|
|
Thema
|
Tipp oder Trick...
|
|
|
Abfrage, ave
(siehe auch doBy)
|
Enthält Ihr z. B. Datensatz (data.frame) eine Gruppierungsvariable (Faktor) und Sie möchten bezogen auf die Ausprägungen der Gruppierungsvariablen eine Funktion, wie z. B. mean() um den Mittelwert zu schätzen, anwenden, können Sie dies über die Funktion ave() erreichen. In dem folgenden Beispieldatensatz Daten ist die Variable x4 die Gruppierungsvariable.
Für die Beobachtung (Variable) x1 soll in Abhängigkeit von x4 der Mittelwert geschätzt werden. D. h., es soll der Mittelwert für die x1-Ausprägungen geschätzt werden wenn x4 = ja und wenn x4 = nein ist:
> # Daten$x1: Daten zur Auswertung, Daten$x4: Daten zur Gruppierung
> ave(Daten$x1, Daten$x4, FUN = mean)
[1] 2.683333 2.683333 3.150000 2.683333 3.150000 3.150000 2.683333 2.683333
[9] 2.683333 3.150000
Der Mittelwert 2.683333 wird für x1 geschätzt, wenn x4 = ja ist und der Mittelwert 3.15 wenn x4 = nein ist.
|
|
|
Abfrage, which
|
Eine hilfreiche Funktion zur Positionsbestimmung bestimmter Merkmalsausprägungen in z. B. einem Datensatz (data.frame) ist die Funktion which(). Das folgende Beispiel zeigt die Funktionsweise von which() und dazu wird ein Datensatz erzeugt, der aus 3 Merkmalen mit jeweils 20 zufällig erzeugten Ausprägungen belegt ist:
> Beispiel <- data.frame( x = runif(20), y= runif(20), rquadrat=runif(20))
> Beispiel
x y rquadrat
1 0.695944964 0.33340255 0.05648682
2 0.335060718 0.78721482 0.10211375
3 0.701246433 0.08936092 0.32834186
4 0.840534915 0.04369990 0.99060051
5 0.882632007 0.51493374 0.71436534
6 0.766589350 0.04808933 0.07897418
7 0.322027628 0.08919613 0.61421645
8 0.042117008 0.68832552 0.49323367
9 0.892709922 0.03483853 0.26909765
10 0.638105145 0.43091092 0.29311086
11 0.554493116 0.75929405 0.47527830
12 0.766329948 0.83179695 0.96407855
13 0.096861294 0.38246252 0.42363210
14 0.748016488 0.61555482 0.84409710
15 0.875217988 0.34498665 0.95382735
16 0.007668596 0.53227877 0.28647352
17 0.934916985 0.26000911 0.54084940
18 0.406965658 0.57286062 0.58137832
19 0.788763390 0.90584668 0.25628689
20 0.976127625 0.56602188 0.87808397
Wir möchten nun beispielhaft wissen, für welche Zeilen das Merkmal rquadrat mit einer Ausprägung > 0,8 belegt ist:
Über die Funktion which() mit obigen Argumenten werden 5 Indizes (4 12 14 15 20) ausgegeben und zur schnelleren Identifizierung wurden sie im obigen data.frame Beispiel gelb markiert.
|

|
|
Argumente einer Funktion
|
Sind Ihnen die Argumente zum Funktionsaufruf nicht mehr geläufig und Sie möchten sich auch nicht durch die Hilfefunktion zur Funktion arbeiten, hilft das Argument args(Funktion) weiter. Die Argumente zur Funktion kontingenz() (Kontingenzanalyse) über die Funktion args() aufgerufen, sind folgende:
Oder für die Funktion rnorm() werden folgende Argumente benötigt:
|

|
|
“...”-Argument
|
Wenn Sie eigene Funktionen schreiben und in dieser Funktion z. B. die argumentreiche Funktion plot() verwenden, besteht bei Ihnen sicher der Wunsch nicht alle für plot() nötige Argumente formal anzugeben. Um dennoch die Möglichkeiten von plot() nutzen zu können, bietet R das ...-Punkte-Argument an.
D. h., Sie können über das ...-Punkte-Argument die nötigen Argumente an die plot()-Funktion weiterleiten. Diese Funktionsweise wird am Beispiel der Funktion mean() gezeigt:
Beinhaltet Ihr Datensatz Daten mindestens ein NA (z. B.: 95 94 96 NA 97 93) liefert folgender Funktionsaufruf dreipunkte(Daten) kein brauchbares Ergebnis. Aber durch das ...-Punkte-Argument liefert dieser Aufruf dreipunkte(Daten, na.rm = T) das richtige Ergebnis 95. Das Argument na.rm = T durch die ...-Punkte zur Funktion mean() “durchgeleitet”!
|

|
|
Cloud, R in der -
Amazon Web Services EC2
|
Ist Ihr Computer-System mit der aktuellen R-Aufgabe überfordert oder Sie möchten es einfach nur entlasten, bietet sich das Auslagern der Aufgabe in Cloud an. Verschiedene Internet-Provider oder auch Amazon bieten Computer-System-Kapazitäten zur Miete an. Amazon macht es dem Cloud-Anfänger besonders leicht, weil die mirco-Cloud-Kapazität für 365 Tage kostenfrei angeboten wird. Das bedeutet, Sie können in Ruhe Cloud-Erfahrung sammeln!
Im Folgenden wird das Einrichten und Nutzen einer R-Umgebung unter Windows in einer Amazon-mirco-Cloud als Übersicht beschrieben. Die Beschreibung und Aussagen, besonders zu Kosten, sind ohne Gewähr!
Wie angedeutet, werden nur die wesentliche Schritte beschrieben und der erste wesentliche Schritt ist das Anlegen eines Kontos über das Amazon Web Service Portal und hierzu benötigen Sie eine Kreditkarte! Wenn Sie sich für die kostenfreien Konfigurationen unter Linux oder Windows entscheiden, fallen Kosten im Centbereich an, die aber wahrscheinlich nie beglichen werden müssen! Die Amazon-Beschreibung zum Anlegen einer Instanz (in diesem Beispiel die Windows-Umgebung) wird zurzeit nur in englischer Sprache angeboten, ist aber recht gut.
Der zweite Schritt im Anlegen einer Instanz, ist die Auswahl des Betriebssystems (Bild 1). Die Betriebssystem, die mit eienm Stern gekennzeichnet sind, sind kostenfrei!
Bild 1
Geben Sie der anzulegenden Instanz einen Namen (hier R-Umgebung) und ebenso für den Schlüssel (hier R-EC2), den Sie zum späteren Zugang benötigen. Nach Continue wird Ihnen die Auswahl zur Bestätigung dargestellt (Bild 2). Unter Type können Sie erkennen, dass es sich um die kostenfreie micro-Instanz handelt!
Bild 2
Mit Launch wird die Instanz angelegt (Bild 3):
Bild 3
Bild 3 zeigt noch weitere Informationen, wie die wichtige IP-Adresse (DNS-Name)! Für dieses Beispiel lautet sie bei Auswahl der Region Irland ec2-54-216-114-224.eu-west-1.compute.amazonaws.com. Legen Sie eine weitere oder neue Instanz an, wird eine weitere oder neue IP-Adresse erzeugt. Diese lange IP-Adresse kann auch gegen eine elastische IP-Adresse ausgetauscht werden. Sie sieht wie eine gewohnte IP-Adresse, z. B. 46.51.189.215, aus und kann der angelegten Instanz zugeordnet werden. Die Adresse (egal welcher Typ) wird noch zur Verbindungsherstellung benötigt!
Amazon empfiehlt die Einstellung der Security Groups, was aber für die ersten Gehversuche nicht unbedingt notwendig ist!
Im nächsten Schritt schaffen wir die Voraussetzung zur Verbindungsherstellung über das Windows-Programm Remotedesktop. Der schnelle Weg ist, indem Sie mit der rechten Maustaste in einen freien Bereich des Instanzübersicht (Bild 3) klicken und einen Remotedektop-Shortcut erstellen (Bild 4):
Bild 4
Im Bild 4 ist der Bereich “Retrieve Password” gelb markiert! Diese Funktion benötigen Sie, um aus dem erzeugten Schlüssel R-EC2 das lesbare Passwort zu erzeugen, das Sie zum Aufbau der Remotedesktop-Verbindung benötigen. Nach dem Ausführen des Remotedesktop-Shortcuts muss das Herstellen der Verbindung noch bestätigen werden und dann wird Ihnen auf Ihrem Computer die Oberfläche der angelegten micro-Instanz zur Nutzung angezeigt (Bild 5):
Bild 5
Wenn dann noch die Amazon-Instanz auf Ihren Computer zugreifen kann (Laufwerkfreigabe im Remotedesktop-Programm!) steht dem Kopieren der R-Installationsdatei nichts mehr im Wege (Bild 6):
Bild 6
Nach dem Kopieren auf die Amazon-Instanz können Sie R wie gewohnt installieren und nutzen (Bild 7):
Bild 7
Fertig! Aber noch einen Hinweis: Beenden Sie die Remotedesktop-Verbindung, wird nicht das laufende R-Programm beendet und auch nicht die laufende Instanz (ist ja auch so gewollt)! Sie können also R “arbeiten” lassen und Ihren Computer runterfahren! R und auch die Amazon-Instanz muss explizit beendet und terminiert werden!
|

|
|
Daten aggregieren
(siehe auch doBy)
|
Die Funktion aggregate() verschafft Ihnen einen zusammenfassenden Überblick über Datenobjekte bezüglich statistischer Parameter. Was sich ein wenig holperig anhört, lässt sich durch ein simples Beispiel beschreiben! Dabei wird auf das R-Datenobjekt ChickWeight zurückgegriffen. Verschaffen wir uns einen Überblick über die Datenstruktur:
> data(ChickWeight)
> Hühner <- ChickWeight
> str(Hühner)
Classes ‘nfnGroupedData’, ‘nfGroupedData’, ‘groupedData’ and 'data.frame': 578 obs. of 4 variables:
$ weight: num 42 51 59 64 76 93 106 125 149 171 ...
$ Time : num 0 2 4 6 8 10 12 14 16 18 ...
$ Chick : Ord.factor w/ 50 levels "18"<"16"<"15"<..: 15 15 15 15 15 15 15 15 15 15 ...
$ Diet : Factor w/ 4 levels "1","2","3","4": 1 1 1 1 1 1 1 1 1 1 ...
- attr(*, "formula")=Class 'formula' length 3 weight ~ Time | Chick
.. ..- attr(*, ".Environment")=<environment: R_EmptyEnv>
- attr(*, "outer")=Class 'formula' length 2 ~Diet
.. ..- attr(*, ".Environment")=<environment: R_EmptyEnv>
- attr(*, "labels")=List of 2
..$ x: chr "Time"
..$ y: chr "Body weight"
- attr(*, "units")=List of 2
..$ x: chr "(days)"
..$ y: chr "(gm)"
Der Datensatz beinhaltet zwei nummerische Variablen: weight (das Huhngewicht) und Time (eine zeitliche Klassifikation - Lebenszeit der Hühner). Über die Lebenszeit der Hühner (Time), möchten wir nun das mittlere Huhngewicht (weight) schätzen:
> aggregate(weight~Time, data = Hühner, FUN = "mean")
Time weight
1 0 41.06000
2 2 49.22000
3 4 59.95918
4 6 74.30612
5 8 91.24490
6 10 107.83673
7 12 129.24490
8 14 143.81250
9 16 168.08511
10 18 190.19149
11 20 209.71739
12 21 218.68889
Das mittler Huhngewicht für die Zeitperiode 12 beträgt 129,24. Die Funktion aggregate() bitte noch einige Möglichkeiten, rufen Sie dazu die Hilfe auf!
|

|
|
Daten aggregieren, doBy
|
Über die Funktionen des Paketes doBy können für gruppierte Beobachtungen elegant summarische statistische Parameter ermittelt werden. Die Bandbreite erstreckt sich von den R-Standardfunktion bis zu eigenen Funktionen. Aber wie immer, ein Beispiel sagt mehr als 1000 Worte:
Für das Beispiel wird der "ChickWeight-datasets" der R-Basic-Installation, umbenannt in "Hühner", verwendet:
> str(Hühner)
Classes ‘nfnGroupedData’, ‘nfGroupedData’, ‘groupedData’ and 'data.frame': 578 obs. of 4 variables:
$ weight: num 42 51 59 64 76 93 106 125 149 171 ...
$ Time : num 0 2 4 6 8 10 12 14 16 18 ...
$ Chick : Ord.factor w/ 50 levels "18"<"16"<"15"<..: 15 15 15 15 15 15 15 15 15 15 ...
$ Diet : Factor w/ 4 levels "1","2","3","4": 1 1 1 1 1 1 1 1 1 1 ...
- attr(*, "formula")=Class 'formula' length 3 weight ~ Time | Chick
.. ..- attr(*, ".Environment")=<environment: R_EmptyEnv>
- attr(*, "outer")=Class 'formula' length 2 ~Diet
.. ..- attr(*, ".Environment")=<environment: R_EmptyEnv>
- attr(*, "labels")=List of 2
..$ x: chr "Time"
..$ y: chr "Body weight"
- attr(*, "units")=List of 2
..$ x: chr "(days)"
..$ y: chr "(gm)"
> head(Hühner) # Schauen wir uns die ersten paar Daten über head an
Grouped Data: weight ~ Time | Chick
weight Time Chick Diet
1 42 0 1 1
2 51 2 1 1
3 59 4 1 1
4 64 6 1 1
5 76 8 1 1
6 93 10 1 1
Time ist als Altersklassifikation zu verstehen. Zum Prüfen der später doBy-Funktion schauen wir uns die Altersklassifizierung "12" an:
> Hühner_T12 <- subset(Hühner, Hühner$Time == "12")
> Hühner_T12
Grouped Data: weight ~ Time | Chick
weight Time Chick Diet
7 106 12 1 1
19 122 12 2 1
31 115 12 3 1
43 102 12 4 1
55 141 12 5 1
67 141 12 6 1
79 146 12 7 1
91 110 12 8 1
102 90 12 9 1
114 89 12 10 1
126 168 12 11 1
138 119 12 12 1
150 71 12 13 1
162 164 12 14 1
174 67 12 15 1
182 54 12 16 1
189 98 12 17 1
203 82 12 19 1
215 77 12 20 1
227 217 12 21 2
239 108 12 22 2
251 127 12 23 2
263 70 12 24 2
275 146 12 25 2
287 136 12 26 2
299 115 12 27 2
311 145 12 28 2
323 134 12 29 2
335 115 12 30 2
347 123 12 31 3
359 159 12 32 3
371 137 12 33 3
383 164 12 34 3
395 201 12 35 3
407 145 12 36 3
419 103 12 37 3
431 128 12 38 3
443 130 12 39 3
455 154 12 40 3
467 155 12 41 4
479 160 12 42 4
491 184 12 43 4
503 127 12 44 4
513 135 12 45 4
525 144 12 46 4
537 148 12 47 4
549 154 12 48 4
561 152 12 49 4
573 155 12 50 4
> mean(Hühner_T12$weight)
[1] 129.2449
> sd(Hühner_T12$weight)
[1] 34.1196
Eine Übersicht über den gesamten Datensatz Hühner über die Time- Klassifikation bezüglich Mittelwert und Standardabweichung für das Hühnergewicht weight wird über die Funktion summaryBy ausgegeben:
> Übersicht <- summaryBy(.~Time, data=Hühner, FUN = c(mean, sd))
> Übersicht
Time weight.mean weight.sd
1 0 41.06000 1.132272
2 2 49.22000 3.688316
3 4 59.95918 4.495179
4 6 74.30612 9.012038
5 8 91.24490 16.239780
6 10 107.83673 23.987277
7 12 129.24490 34.119600
8 14 143.81250 38.300412
9 16 168.08511 46.904079
10 18 190.19149 57.394757
11 20 209.71739 66.511708
12 21 218.68889 71.510273
Siehe Zeile 7 und vergleiche mit mean und sd weiter oben!
Daneben Time weight die einzige kontinuierliche Variable ist, wird der .-Operator nur für weight interpretiert. Die Formel-Ausprägung zeigt das gleiche Resultat:
> Übersicht <- summaryBy(weight~Time, data=Hühner, FUN = c(mean, sd))
|

|
|
Daten selektieren und bearbeiten mit dplyr
|
Das Paket dplyr bietet hilfreiche Funktionen zur Datenselektion und -bearbeitung an. Die Entwickler hatten das Ziel, dplyr-Funktionen aus Sicht des Anwenders zu entwickeln und dadurch erscheinen sie möglicherweise einfacher.
Hier finden Sie ein Einführungsvideo zum dplyr-Paket!
|

|
|
Daten selektieren und bearbeiten mit data.table
|
Das data.table-Paket ist ein "altes" und der R-Gemeinde etabliertes Paket zur Datenselektion und -bearbeitung. Das Paket wurde designet, um große Datenmengen schnell und effektiv zu bearbeiten.
Hier finden Sie ein Einführungsvideo zum data.table-Paket!
|

|
|
Daten bereinigen,
Duplikate entfernen
|
Sie verfügen z. B. über einen Datensatz (data.frame), der doppelte Einträge enthält und diese möchten Sie vor der weiteren Analyse entfernen. Hier hilft die Funktion duplicated() weiter! Hier ein Beispiel:
> Daten # Duplikate sind gelb markiert!
y x1 x2 x3
1 1.0 0.5 0.03 0.20
2 2.1 1.2 0.07 0.45
3 3.1 1.5 0.13 0.60
4 3.1 1.5 0.13 0.60
5 3.9 2.1 0.22 0.80
6 5.0 2.6 0.30 1.00
7 5.8 3.0 0.32 1.20
8 5.8 3.0 0.32 1.20
9 7.3 3.6 0.41 1.50
10 8.1 4.1 0.46 1.76
11 9.2 4.6 0.55 1.90
12 10.3 5.5 0.62 2.10
# Entfernen der Duplikate über duplicated():
> Daten_einmalig <- Daten[!duplicated(Daten),]
> Daten_einmalig
y x1 x2 x3
1 1.0 0.5 0.03 0.20
2 2.1 1.2 0.07 0.45
3 3.1 1.5 0.13 0.60
5 3.9 2.1 0.22 0.80
6 5.0 2.6 0.30 1.00
7 5.8 3.0 0.32 1.20
9 7.3 3.6 0.41 1.50
10 8.1 4.1 0.46 1.76
11 9.2 4.6 0.55 1.90
12 10.3 5.5 0.62 2.10
Die “fehlenden Indizes 4 und 8 zeigen, wo sich vor dem Entfernen die Duplikate befunden haben!
|

|
|
Dateien einlesen
|
Möchten Sie dem Anwender einer Funktion die Auswahl der zu ladenden Datei überlassen, hilft Ihnen die Funktion choose.files() weiter. Wird die Funktion aufgerufen, wird das vom Betriebssystem abhängige Dateiauswahlmenü gezeigt:
Folgenden R-Code könnten Sie in Ihrer Funktion nun nutzen:
Datei <- choose.files(filters = Filters[c("txt", "All"),])
Inhalt <- read.table(Datei, header = T, sep=";")
Durch obige Filtereinstellung (filters), werden nur “.txt”-Dateien angezeigt. Diese Einstellung kann natürlich geändert werden:
Die Variable Datei enthält nach der Auswahl die komplette Pfad-Angabe und nach dem Einlesen der Datei in das Objekt Inhalt könnte die Ausgabe wie folgt aussehen:
Eine Variante zum z. B. eine Tabellenkalkulationsdatei einzulesen ist:
> Daten <- read.csv2(choose.files(), ...)
Die ... sind Platzhalter für die read.csv2-Funktionsargumente.
|

|
|
Daten einlesen mit dem ff-Paket,
Thema “BigData”
|
Mit den Funktionen des ff-Paketes können sogenannte BigData-Dateien sequenziell in die R-Umgebung geladen werden. Das Paket bietet viele Funktionen, die nicht nur des Datenladens dienen, sondern eine direkte Datenbearbeitung ermöglichen. Wegen der Vielfältigkeit der Möglichkeiten für diese Funktionen, wird allerdings auf die Dokumentation zum Paket verwiesen.
In dieser Beschreibung wird unter einer BigData-Datei eine so große Datei verstanden, dass sie nicht mehr in den Speicher des Ihnen zur Verfügung stehenden Computers geladen werden kann verstanden.
Die folgenden Beispiele sind überschaubar und es werden jeweils nur 100 Zeilen einer kleinen Datei eingelesen. Die Beispieldatei ist weit entfernt vom "BigData-Datei"-Anspruch.
Über die Funktion read.csv2.ffdf() werden die ersten 100 Zeilen der Datei Alle_Weindaten.csv ( P. Cortez, A. Cerdeira, F. Almeida, T. Matos and J. Reis, Modeling wine preferences by data mining from physicochemical properties, In Decision Support Systems, Elsevier, 47(4):547-553. ISSN: 0167-9236.) eingelesen und in dem ff-Objekt TeilMenge abgelegt:
> TeilMenge <- read.csv2.ffdf(file = "Alle_Weindaten.csv", header = TRUE, nrows = 100)
> str(TeilMenge)
List of 3
$ virtual: 'data.frame': 13 obs. of 7 variables:
.. $ VirtualVmode : chr "integer" "double" "double" "double" ...
.. $ AsIs : logi FALSE FALSE FALSE FALSE FALSE FALSE ...
.. $ VirtualIsMatrix : logi FALSE FALSE FALSE FALSE FALSE FALSE ...
.. $ PhysicalIsMatrix : logi FALSE FALSE FALSE FALSE FALSE FALSE ...
.. $ PhysicalElementNo: int 1 2 3 4 5 6 7 8 9 10 ...
.. $ PhysicalFirstCol : int 1 1 1 1 1 1 1 1 1 1 ...
.. $ PhysicalLastCol : int 1 1 1 1 1 1 1 1 1 1 ...
.. - attr(*, "Dim")= int 100 13
.. - attr(*, "Dimorder")= int 1 2
$ physical: List of 13
.. $ Type : list()
.. ..- attr(*, "physical")=Class 'ff_poi... Ausgabe aus Darstellungsgründen abgebrochen!
Lassen Sie Sie sich den Inhalt des Objektes auf der Konsole anzeigen, werden Ihnen neben reichlich Objektinformationen die ersten und letzen Datenzeilen des Datensatzes angezeigt:
> TeilMenge
ffdf (all open) dim=c(100,13), dimorder=c(1,2) row.names=NULL
ffdf virtual mapping
PhysicalName VirtualVmode PhysicalVmode ... Ausgabe aus Darstellungsgründen gekürzt!
Type FixedAcidity VolatileAcidity CitricAcid ResidualSugar Chlorides FreeSulfurDioxide
1 r 7.4 0.700 0.00 1.9 0.076 11
2 r 7.8 0.880 0.00 2.6 0.098 25
3 r 7.8 0.760 0.04 2.3 0.092 15
4 r 11.2 0.280 0.56 1.9 0.075 17
5 r 7.4 0.700 0.00 1.9 0.076 11
6 r 7.4 0.660 0.00 1.8 0.075 13
7 r 7.9 0.600 0.06 1.6 0.069 15
8 r 7.3 0.650 0.00 1.2 0.065 15
: : : : : : : :
93 r 8.6 0.490 0.29 2.0 0.110 19
94 r 7.7 0.490 0.26 1.9 0.062 9
95 r 5.0 1.020 0.04 1.4 0.045 41
96 r 4.7 0.600 0.17 2.3 0.058 17
97 r 6.8 0.775 0.00 3.0 0.102 8
98 r 7.0 0.500 0.25 2.0 0.070 3
99 r 7.6 0.900 0.06 2.5 0.079 5
100 r 8.1 0.545 0.18 1.9 0.080 13
... Ausgabe aus Darstellungsgründen gekürzt!
Auf den Inhalt der TeilMenge kann mit den bekannten R-Anweisungen zugegriffen werden:
> TeilMenge[1:3,]
Type FixedAcidity VolatileAcidity CitricAcid ResidualSugar Chlorides FreeSulfurDioxide
1 r 7.4 0.70 0.00 1.9 0.076 11
2 r 7.8 0.88 0.00 2.6 0.098 25
3 r 7.8 0.76 0.04 2.3 0.092 15
... Ausgabe aus Darstellungsgründen gekürzt!
Auf die nächsten 100 Elemente kann über das Argument skip = 100 zugegriffen werden. Allerdings wird hier auf das Auslesen der Kopfzeile mit header = FALSE verzichtet:
> TeilMenge2 <- read.csv2.ffdf(file = "Alle_Weindaten.csv", header = FALSE, skip = 100, nrows = 100)
> TeilMenge2[1:2,]
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13
1 r 8.1 0.545 0.18 1.9 0.080 13 35 0.9972 3.3 0.59 9.0 6
2 r 8.3 0.610 0.30 2.1 0.084 11 50 0.9972 3.4 0.61 10.2 6
Auf diese Art und Weise kann die BigData-Datei sequenziell analysiert werden. Mit der read-Funktion sind Sie über das Argument skip= 1 + nrow in der Lage, sich sequentiell durch die Datei zu "arbeiten".
|

|
|
Daten einlesen, aus dem Internet (url)
|
Über die read-Funktion können Sie Daten aus dem Internet in die R-Umgebung laden. Dazu bietet die read-Funktion diverse Protokolle an und in diesem Beispiel wird die Datei Eurostat_tsdec420.csv über das http-Protokoll in die R-Umgebung geladen. Diese Tabelle (Code tsdec420) beschreibt die Beschäftigungsrate in der EU für die Geschlechter männlich/weiblich (Darstellungssicht des EU-Servers):
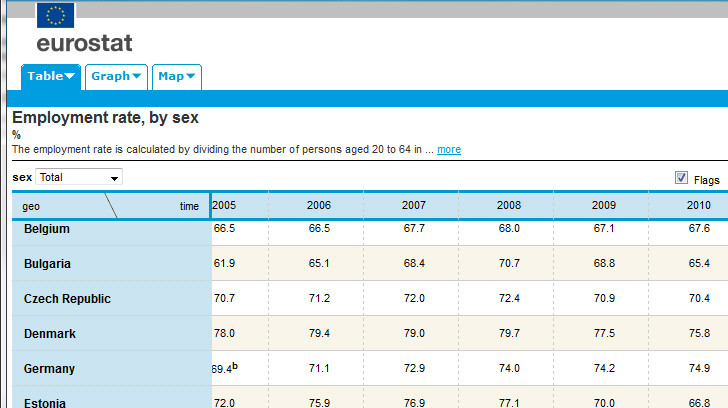
Diese Datei können Sie als csv-Datei über die folgende Variante der read-Funktion auf Ihren Computer laden:
> EuroDaten <- read.csv2("http://www.r-statistik.de/R_erweitern/Tipps_Tricks/Eurostat_tsdec420.csv")
Die Prüfung mit str() sollte folgende Ausgabe zeigen:
> str(EuroDaten)
'data.frame': 39 obs. of 23 variables:
$ geo.time: Factor w/ 39 levels "Belgien","Bulgarien",..: 8 7 10 9 1 2 34 3 4 6 ...
$ X1992 : num NA NA NA NA 61.3 NA NA 75.7 68.9 NA ...
$ X1993 : num NA NA NA NA 60.8 NA NA 74 67.7 NA ...
$ X1994 : num NA NA NA NA 60.7 NA NA 74.1 67.5 NA ...
$ X1995 : num NA NA NA NA 61.2 NA NA 75 67.6 NA ...
$ X1996 : num NA NA NA NA 61.4 NA NA 75.5 67.2 NA ...
$ X1997 : num NA 65.1 62.9 62.9 62.1 NA NA 76.4 66.9 NA ...
$ X1998 : num NA 65.5 63.5 63.5 62.7 NA 73.4 76.8 67.2 71.2 ...
$ X1999 : num NA 66.2 64.6 64.6 64.5 NA 71.5 77.7 68.3 68.5 ...
$ X2000 : num NA 66.6 65.5 65.5 65.8 55.3 71 78 68.8 66.6 ...
$ X2001 : num NA 66.9 66.2 66.2 65 54.8 71.2 78.3 69.1 67.4 ...
$ X2002 : num 66.7 66.7 66.5 66.5 65 55.8 71.6 77.7 68.8 68 ...
$ X2003 : num 67 67.1 66.9 66.9 64.7 58 70.7 77.3 68.4 69.6 ...
$ X2004 : num 67.4 67.4 67.3 67.3 65.6 60.1 70.1 77.6 68.8 70.3 ...
$ X2005 : num 67.9 68 67.9 67.9 66.5 61.9 70.7 78 69.4 72 ...
$ X2006 : num 68.9 69 69 68.9 66.5 65.1 71.2 79.4 71.1 75.9 ...
$ X2007 : num 69.8 69.9 69.9 69.8 67.7 68.4 72 79 72.9 76.9 ...
$ X2008 : num 70.3 70.3 70.2 70.2 68 70.7 72.4 79.7 74 77.1 ...
$ X2009 : num 69 69 68.8 68.8 67.1 68.8 70.9 77.5 74.2 70 ...
$ X2010 : num 68.5 68.6 68.4 68.4 67.6 65.4 70.4 75.8 74.9 66.8 ...
$ X2011 : num 68.5 68.6 68.5 68.5 67.3 62.9 70.9 75.7 76.5 70.6 ...
$ X2012 : num 68.4 68.5 68 68 67.2 63 71.5 75.4 76.9 72.2 ...
$ X2013 : num 68.4 68.5 67.7 67.7 67.2 63.5 72.5 75.6 77.3 73.3 ...
|

|
|
Daten, data.frame
head() und tail()
|
Möchten Sie sich einen schnellen Überblick über einen Datensatz (Datenrahmen, data.frame) verschaffen, können die Funktionen head() und tail() hilfreich sein. Über diese Funktionen werden Ihen jeweils die ersten 6 bzw. letzten 6 Datensätze auf der Konsole ausgegeben:
Für beide Funktion ist die Ausgabe von 6 Datensätzen die Standardeinstellung. Über das Argument n können Sie eine beliebige Anzahl Datensätze ausgeben, z. B. 10 Datensätze: head(cars, n = 10).
|

|
|
Daten ergänzen, with()
|
Sie möchten ein Merkmal eines Datensatzes (data.frame) transformieren, z. B. potenzieren, und dieses transformierte Merkmal direkt in den Datensatz aufnehmen, hilft die Funktion with() weiter:
> Daten # Ausgangsdaten
y x1 x2 x3
1 1.0 0.5 0.03 0.20
2 2.1 1.2 0.07 0.45
3 3.1 1.5 0.13 0.60
4 3.9 2.1 0.22 0.80
5 5.0 2.6 0.30 1.00
6 5.8 3.0 0.32 1.20
7 7.3 3.6 0.41 1.50
8 8.1 4.1 0.46 1.76
9 9.2 4.6 0.55 1.90
10 10.3 5.5 0.62 2.10
# data.frame mit x3^2 ergänzen:
> Daten$x_quadrat <- with(Daten, x3^2)
> Daten
y x1 x2 x3 x_quadrat
1 1.0 0.5 0.03 0.20 0.0400
2 2.1 1.2 0.07 0.45 0.2025
3 3.1 1.5 0.13 0.60 0.3600
4 3.9 2.1 0.22 0.80 0.6400
5 5.0 2.6 0.30 1.00 1.0000
6 5.8 3.0 0.32 1.20 1.4400
7 7.3 3.6 0.41 1.50 2.2500
8 8.1 4.1 0.46 1.76 3.0976
9 9.2 4.6 0.55 1.90 3.6100
|

|
|
Daten ergänzen,
transform()
|
Die Funktion transform() transformiert Daten in eine andere Form. Was bedeutet das? Das wird am Beispiel des airquality-Datensatzes erklärt:
> Daten <- airquality
> str(Daten)
'data.frame': 153 obs. of 6 variables:
$ Ozone : int 41 36 12 18 NA 28 23 19 8 NA ...
$ Solar.R: int 190 118 149 313 NA NA 299 99 19 194 ...
$ Wind : num 7.4 8 12.6 11.5 14.3 14.9 8.6 13.8 20.1 8.6 ...
$ Temp : int 67 72 74 62 56 66 65 59 61 69 ...
$ Month : int 5 5 5 5 5 5 5 5 5 5 ...
$ Day : int 1 2 3 4 5 6 7 8 9 10 …
> Daten
Ozone Solar.R Wind Temp Month Day
1 41 190 7.4 67 5 1
2 36 118 8.0 72 5 2
3 12 149 12.6 74 5 3
4 18 313 11.5 62 5 4
5 NA NA 14.3 56 5 5
6 28 NA 14.9 66
Der Datensatz beinhaltet die Beobachtung Temp in Grad Fahrenheit. Diese Temperatur wird in Grad Celsius transformieren und als Objekt Temp_C zum Datensatz Daten addiert:
> Daten <- transform(airquality, Temp_C = round((Temp-32)/1.8, digits = 1))
> str(Daten)
'data.frame': 153 obs. of 7 variables:
$ Ozone : int 41 36 12 18 NA 28 23 19 8 NA ...
$ Solar.R: int 190 118 149 313 NA NA 299 99 19 194 ...
$ Wind : num 7.4 8 12.6 11.5 14.3 14.9 8.6 13.8 20.1 8.6 ...
$ Temp : int 67 72 74 62 56 66 65 59 61 69 ...
$ Month : int 5 5 5 5 5 5 5 5 5 5 ...
$ Day : int 1 2 3 4 5 6 7 8 9 10 ...
$ Temp_C : num 19.4 22.2 23.3 16.7 13.3 18.9 18.3 15 16.1 20.6 …
> Daten
Ozone Solar.R Wind Temp Month Day Temp_C
1 41 190 7.4 67 5 1 19.4
2 36 118 8.0 72 5 2 22.2
3 12 149 12.6 74 5 3 23.3
4 18 313 11.5 62 5 4 16.7
5 NA NA 14.3 56 5 5 13.3
6 28 NA 14.9 66 5 6 18.9
7 23 299 8.6 65 5 7 18.3
8 19 99 13.8 59 5 8 15.0
9 8 19 20.1 61 5 9 16.1
10 NA 194
|

|
|
Daten, fehlende Werte (NA’s)
|
Ihre Beobachtungen (Datensätze, Merkmale, ...) beinhalten unter Umständen fehlende Werte, so genannte NA’s. Auch wenn viele R-Funktionen interne Routine zum Umgang mit NA’s haben, sollten Sie sich immer überlegen, wie Sie mit fehlenden Werten umgehen. Diese Überlegung kann ich Ihnen an dieser Stelle nicht abnehmen, aber einige Möglichkeiten in dem R-seitigen Umgang mit NA’s in R beschreiben. Als Beispieldatensatz wird der Datensatz mtcars, abgelegt in carData, verwendet. Der Umgang mit fehlenden Werten wird auch im Video R und NA’s beschrieben.
Sie importieren Daten aus einem anderen Statistikprogramm über eine csv-Datei in die R-Umgebung. Diese Daten beinhalten fehlende Werte, die als -99 kodiert sind. Damit R sie erkennt, soll -99 in NA umkodiert werden. Hier die Sicht auf die Daten über den Dateneditor (Abb. 1):
Abb. 1, die manipulierten mtcars-Daten
Über folgende R-Anweisungen werden für carData$drat die Ausprägung -99 gegen NA’s ausgetauscht:
> is.na(carData$drat) <- which(carData$drat == -99)
# Prüfen, ob der Austausch erfolgreich war:
> str(carData)
'data.frame': 32 obs. of 11 variables:
$ mpg : num 21 21 22.8 21.4 18.7 18.1 14.3 24.4 22.8 19.2 ...
$ cyl : num 6 6 4 6 8 6 8 4 4 6 ...
$ disp: num 160 160 108 258 360 ...
$ hp : num 110 110 93 110 175 105 245 62 95 123 ...
$ drat: num 3.9 3.9 NA 3.08 3.15 NA NA 3.69 3.92 3.92 ...
$ wt : num 2.62 2.88 2.32 3.21 3.44 ...
$ qsec: num 16.5 17 18.6 19.4 17 ...
$ vs : num 0 0 1 1 0 1 0 1 1 1 ...
$ am : num 1 1 1 0 0 0 0 0 0 0 ...
$ gear: num 4 4 4 3 3 3 3 4 4 4 ...
$ carb: num 4 4 1 1 2 1 4 2 2 4 ...
Sie können die NA’s natürlich auch gegen Werte austauschen. In dem folgenden Beispiel, werden die NA’s gegen -99 ausgetauscht (ist vielleicht nicht das beste Beispiel):
> carData[is.na(carData$drat),"drat"] <- -99
# Prüfen, ob der Austausch erfolgreich war:
> str(carData)
'data.frame': 32 obs. of 11 variables:
$ mpg : num 21 21 22.8 21.4 18.7 18.1 14.3 24.4 22.8 19.2 ...
$ cyl : num 6 6 4 6 8 6 8 4 4 6 ...
$ disp: num 160 160 108 258 360 ...
$ hp : num 110 110 93 110 175 105 245 62 95 123 ...
$ drat: num 3.9 3.9 -99 3.08 3.15 -99 -99 3.69 3.92 3.92 ...
$ wt : num 2.62 2.88 2.32 3.21 3.44 ...
$ qsec: num 16.5 17 18.6 19.4 17 ...
$ vs : num 0 0 1 1 0 1 0 1 1 1 ...
$ am : num 1 1 1 0 0 0 0 0 0 0 ...
$ gear: num 4 4 4 3 3 3 3 4 4 4 ...
$ carb: num 4 4 1 1 2 1 4 2 2 4 ...
Da der Umgang mit fehlenden Werten immer ein wenig kritisch ist, können auch alle Datensätze mit fehlenden Werten mit der Funktion na.exclude(Daten) aus dem Datensatz entfernt werden! D. h., alle Zeilen, die NA’s beinhalten, werden aus dem Datensatz entfernt:
> carData <- na.exclude(carData)
# Prüfen, ob der Austausch erfolgreich war:
> str(carData)
'data.frame': 28 obs. of 11 variables:
$ mpg : num 21 21 21.4 18.7 24.4 19.2 17.8 16.4 17.3 15.2 ...
$ cyl : num 6 6 6 8 4 6 6 8 8 8 ...
$ disp: num 160 160 258 360 147 ...
$ hp : num 110 110 110 175 62 123 123 180 180 180 ...
$ drat: num 3.9 3.9 3.08 3.15 3.69 3.92 3.92 3.07 3.07 3.07 ...
$ wt : num 2.62 2.88 3.21 3.44 3.19 ...
$ qsec: num 16.5 17 19.4 17 20 ...
$ vs : num 0 0 1 0 1 1 1 0 0 0 ...
$ am : num 1 1 0 0 0 0 0 0 0 0 ...
$ gear: num 4 4 3 3 4 4 4 3 3 3 ...
$ carb: num 4 4 1 2 2 4 4 3 3 3 ...
- attr(*, "na.action")=Class 'exclude' Named int [1:4] 3 6 7 9
.. ..- attr(*, "names")= chr [1:4] "Datsun 710" "Valiant" "Duster 360" "Merc 230"
Ihnen fällt auf, dass der Datensatz 4 Zeilen weniger enthält als die str()-Ausgabe weiter oben? Vergleichen Sie Abb. 2 mit Abb.1 und Ihnen wird auffallen, dass die Zeilen mit NA fehlen (Abb. 2):
Abb. 2, Zeilen mit NA’s wurden entfernt
|

|
|
Daten unter dem Mengenaspekt
|
Sie möchten den Inhalt zweier Vektoren unter den Begriffen der Mengenlehre vergleichen und behandeln? Dann helfen Ihnen die Funktionen intersect(), union(), setdiff(), und setequal() weiter:
- Durchschnitt (gleiche Werte in a und b):
> a <- c(1,2,3,4,5,6,7,8,9)
> b <- c(5,6,9,10,12)
> intersect(a, b)
[1] 5 6 9
- Vereinigung von a und b:
> union(a, b)
[1] 1 2 3 4 5 6 7 8 9 10 12
- Differenz von a und b:
> setdiff(a, b)
[1] 1 2 3 4 7 8
- Gleichheitsprüfung beider Mengen (Vektoren):
> setequal(a, b)
[1] FALSE
|

|
|
Daten recodieren,
recode()
|
Ist es nötig, z. B. in einem Datensatz die Ausprägung eines Faktor-Merkmals (z. B. mit der Ausprägung ja und nein) in eine numerische Ausprägung umzuwandeln, können Sie dies elegant mit der Funktion recode aus dem car-Paket erledigen. In dem folgenden Beispiel soll zur Ausprägung ja/nein des Merkmals x4 des folgenden Datensatzes Daten eine neues Merkmal aufgenommen werden, dass in Abhängig zur Ausprägung von x4 ein entsprechendes numerisches Datum enthält:
> Daten # Ausgangsdaten
y x1 x2 x3 x4
1 1.0 0.5 0.03 0.20 ja
2 2.1 1.2 0.07 0.45 ja
3 3.1 1.5 0.13 0.60 nein
4 3.9 2.1 0.22 0.80 ja
5 5.0 2.6 0.30 1.00 nein
6 5.8 3.0 0.32 1.20 nein
7 7.3 3.6 0.41 1.50 ja
8 8.1 4.1 0.46 1.76 ja
9 9.2 4.6 0.55 1.90 ja
10 10.3 5.5 0.62 2.10 nein
> # x4 für “ja” in 1 und für “nein” in 0 wandeln und das Ergebnis als neues Merkmal
> x4_num aufnehmen:
> Daten$x4_num <- recode(Daten$x4, '"ja" = 1; "nein" = 0', as.factor = FALSE)
> Daten
y x1 x2 x3 x4 x4_num
1 1.0 0.5 0.03 0.20 ja 1
2 2.1 1.2 0.07 0.45 ja 1
3 3.1 1.5 0.13 0.60 nein 0
4 3.9 2.1 0.22 0.80 ja 1
5 5.0 2.6 0.30 1.00 nein 0
6 5.8 3.0 0.32 1.20 nein 0
7 7.3 3.6 0.41 1.50 ja 1
8 8.1 4.1 0.46 1.76 ja 1
9 9.2 4.6 0.55 1.90 ja 1
10 10.3 5.5 0.62 2.10 nein 0
Sie können natürlich auch Daten$x4 mit dem gewandelten Wert überschreiben, empfohlen wird dies aber nicht!
|

|
|
Daten, Tabellen mit ftable
|
Kontigenztabellen / Kreuztabellen zeigen Beziehungen zwischen Daten. Ihnen ist sicher die Funktion table() bekannt, aber deren Ausgabe kann unter Umständen unübersichtlich sein. Ein Beispiel dazu ist der Datensatz Titanic:
> str(Titanic)
table [1:4, 1:2, 1:2, 1:2] 0 0 35 0 0 0 17 0 118 154 ...
- attr(*, "dimnames")=List of 4
..$ Class : chr [1:4] "1st" "2nd" "3rd" "Crew"
..$ Sex : chr [1:2] "Male" "Female"
..$ Age : chr [1:2] "Child" "Adult"
..$ Survived: chr [1:2] "No" "Yes"
Lassen Sie sich die obige Tabelle auf dem Bildschirm ausgeben, benötigen Sie ein wenig Zeit, um einen Überblick zu erhalten! Die Funktion ftable() macht aus dieser komplexeren Tabelle eine flache Tabelle:
> ftable(Titanic)
|
|
|
|
Survived
|
No
|
Yes
|
|
Class
|
Sex
|
Age
|
|
|
|
|
1st
|
Male
|
Child
|
|
0
|
5
|
|
|
|
Adult
|
|
118
|
57
|
|
|
Female
|
Child
|
|
0
|
1
|
|
|
|
Adult
|
|
4
|
140
|
|
2nd
|
Male
|
Child
|
|
0
|
11
|
|
|
|
Adult
|
|
154
|
14
|
|
|
Female
|
Child
|
|
0
|
13
|
|
|
|
Adult
|
|
13
|
80
|
|
3rd
|
Male
|
Child
|
|
35
|
13
|
|
|
|
Adult
|
|
387
|
75
|
|
|
Female
|
Child
|
|
17
|
14
|
|
|
|
Adult
|
|
89
|
76
|
|
Crew
|
Male
|
Child
|
|
0
|
0
|
|
|
|
Adult
|
|
670
|
192
|
|
|
Female
|
Child
|
|
0
|
0
|
|
|
|
Adult
|
|
3
|
20
|
Weiter Informationen, z. B. zur Variablen-Selektion, erhalten Sie über die Hilfe-Funktion.
|

|
|
Daten, transformieren in wide/long-Format
|
Die Daten liegen im "wide"-Format vor, d.h., die Messpunkte MP1 bis MP4 die hier als Beispiel gewählt wurden, befinden sich in einer Datenzeile:
Durch die Funktion gather des tidyr-Paketes können die Daten über die Messpunkte MP1 bis MP4 in das "long"-Format, d. h., pro Messpunkt eine Datenzeile, transformiert werden:
> Daten_long <- gather(Daten_wide, key = MP, value = value, MP1:MP4)
> Daten_long
Merkmal MP value
1 A MP1 10.00
2 B MP1 30.20
3 C MP1 0.98
4 A MP2 10.30
5 B MP2 28.90
6 C MP2 1.02
7 A MP3 9.80
8 B MP3 29.50
9 C MP3 1.03
10 A MP4 10.40
11 B MP4 30.50
12 C MP4 1.01
In diesem Format sind dann auch zusammenfassende Analysen wieder möglich. Hier wird der Mittelwert für die drei Merkmalsausprägungen A, B und C geschätzt:
Der Rückweg vom "long"-Format ins "wide"-Format erfolgt über die Funktion spread statt:
|

|
|
Daten konvertieren reshape2, melt
|
Über das Paket reshape2 können Datenobjekte “redesigned” werden. Im Folgenden Beispiel wird das Datenobjekt list in einem data.frame-Objekt gewandelt:
> Liste
[[1]]
[1] 11 22 33 44 55 66
[[2]]
[1] 111 222 333 444 555 666
[[3]]
[1] "A" "B" "C" "E" "F" "G"
> Liste_DF <- melt(Liste)
> Liste_DF
value L1
1 11 1
2 22 1
3 33 1
4 44 1
5 55 1
6 66 1
7 111 2
8 222 2
9 333 2
10 444 2
11 555 2
12 666 2
13 A 3
14 B 3
15 C 3
16 E 3
17 F 3
18 G 3
> str(Liste_DF)
'data.frame':17 obs. of 2 variables:
$ value: chr "11" "22" "33" "44" ...
$ L1 : int 1 1 1 1 1 1 2 2 2 2 ...
Das ist natürlich nicht die wesentliche Qualität des Paketes, sondern die Aggregierungs-Funktion cast() (genau: acast oder dcast) die nach melt() auf Datenobjekt angewendet werden kann. Schauen Sie sich das Paketbeispiel einfach mal an.
|
|
|
Daten, Zeichenketten bearbeiten
|
Das Paket stringr beinhaltet zahlreiche effektive Funktionen zur Zeichenketten(String-)bearbeitung. Die folgenden kleinen Beispiele sollen einen Eindruck über die Möglichkeiten des Paketes vermitteln:
- Konstruieren wir 2 Zeichenketten:
- Über die Funktion length() erhalten wir das bekannte Ergebnis, dass 1 Element in den Objekten a und b enthalten ist:
Um einzelne Zeichen der Zeichenkette zu bearbeiten, hilft uns diese Funktion nicht nicht weiter. Hier kommen die Funktionen des Paketes stringr zum Zuge:
- Über die Funktion str_length() des Paketes, erhalten wir die Anzahl der Zeichen in den Zeichenketten:
- Über die Funktion str_extract_all() kann nun die Zeichenkette zerlegt werden. Die Zeichenkette wird als Liste ausgegeben:
> str_extract_all(a, regex("[a-z]", TRUE))
[[1]]
[1] "E" "r" "s" "t" "e" "Z" "e" "i" "c" "h" "e" "n" "k" "e" "t" "t" "e"
> str_extract_all(b, regex("[a-z]", TRUE))
[[1]]
[1] "Z" "w" "e" "i" "t" "e" "Z" "e" "i" "c" "h" "e" "n" "k" "e" "t" "t" "e”
- Auf zum Beispiel das 2. Zeichen der Zeichenkette a kann über Listen-Argumente zugegriffen werden:
- Auch können Sub-Zeichenketten innerhalb der Zeichenkette elegant lokalisiert werden:
Das Paket bietet wie erwähnt, noch weitere hilfreiche Funktionen zur Zeichenkettenanalyse und -bearbeitung.
|

|
|
Daten, Zeichenketten trimmen
|
Die Auswertung bzw. Bearbeitungen von Texten kann es erforderlich mache, führende oder abschließende Leerzeichen zu entfernen. Eine einfache und schnelle Methode dazu ist trimws():
> text <- " Mein Text! " # Beispieltext
> trimws(text)
[1] "Mein Text!"
Als Variante, kann auch links oder rechts getrimmt werden:
> trimws(text, "l")
[1] "Mein Text! "
> trimws(text, "r")
[1] " Mein Text!"
|

|
|
Datenobjekt-Info
|
Das Paket Hmisc bietet einige nützliche Funktionen an und eine davon ist die Funktion describe(). Über diese Funktion werden Informationen zum R-Datenobjekt, z. B. eines Datensatzes (data.frame()) ausgegeben. Hier ein Beispiel zu einem Datensatz:
Und hier die Information über die Funktion describe():
> describe(Daten)
Daten
5 Variables 9 Observations
-----------------------------------------------------------------------------
Wirkstoff.A
n missing unique Mean
9 0 8 99.17
98.8 98.9 99 99.1 99.2 99.3 99.4 99.5
Frequency 1 1 1 1 1 2 1 1
% 11 11 11 11 11 22 11 11
-----------------------------------------------------------------------------
Wirkstoff.B
n missing unique Mean
9 0 7 97.19
96.9 97 97.1 97.2 97.3 97.4 97.5
Frequency 2 1 1 1 1 2 1
% 22 11 11 11 11 22 11
-----------------------------------------------------------------------------
Wirkstoff.C
n missing unique Mean
9 0 7 95.46
95.1 95.3 95.4 95.5 95.6 95.7 95.8
Frequency 2 1 1 1 2 1 1
% 22 11 11 11 22 11 11
--------------------------------------------------------------------------------
Pruefer
n missing unique
9 0 2
Karl (4, 44%), Peter (5, 56%)
-----------------------------------------------------------------------------
Datum
n missing unique
9 0 9
2008-08-10 2008-08-11 2008-08-12 2008-08-13 2008-08-14 2008-08-15
Frequency 1 1 1 1 1 1
% 11 11 11 11 11 11
2008-08-16 2008-08-17 2008-08-18
Frequency 1 1 1
% 11 11 11
-----------------------------------------------------------------------------
|

|
|
Datenobjekt-Info anlegen
|
Über die Funktion comment() sind Sie in der Lage, einer z. B. kryptischen Variablenbezeichnung einen erklärenden Kommentar anzuheften. Hierzu ein kleines Bespiel:
Der obige Kommentar “Mein Kommentar” wurde dem Objekt x zugeordnet. Lassen Sie sich wie gewohnt den Inhalt von x anzeigen oder verwenden ihn in Berechnungen, merken Sie davon nichts. Sie können sich den Kommentar mit comment(x) oder auch über str() anzeigen lassen:
> comment(x)
[1] "Mein Kommentar"
> str(x)
atomic [1:100] -0.0776 0.7635 0.5122 -0.8217 -0.9334 ...
- attr(*, "comment")= chr "Mein Kommentar"
Die Erweiterung desObjektes x lässt sich auch über die Funktion attributes() zeigen:
|

|
|
Datenbank ODBC
CSV
|
Datenbankanbindung über ODBC und dem Paket RODBC
In diesem Beispiel wird die Datenbankanbindung einer Excel-Datei beschrieben. Ob Sie diesen Dateityp als “Datenbank” ansehen, soll nicht diskutiert werden. In diesem Beispiel geht es um die Vorgehensweise über ODBC Zugriff auf eine Datei zu erhalten.
Bevor unter dem Betriebssystem MS-Windows© 7 auf eine Excel-Datei über ODBC zugegriffen werden kann, muss über den ODBC-Datenquellen-Adiminstrator eine Verbindung zu diesem Dateityp installiert werden (In den allermeisten Fällen ist dieser Schritt nicht notwenig, da Excel als Datenquelle schon bekannt sein sollte!). Dazu rufen Sie wie in Bild 1 gezeigt Programm Datenquellen (ODBC) auf:
Bild 1
Im Bereich Benutzer-DSN prüfen wir den Eintrag der Excel Files als Datenquelle (Bild 2):
Bild 2
Sollte der Eintrag, insbesondere für Datenbanken, nicht vorhanden sein, muss der Treiber der Datenbank vor dem Aufruf des ODBC-Datenquellen-Adiminstrator installiert werden. Die Datenquelle kann dann über die Schaltfläche Hinzufügen zugefügt werden.
Schließen Sie die ODBC-Datenquellen-Adiminstration ab und laden in die R-Umgebung (nur 32 Bit) das Paket RODBC. Um nun mit der Datenbank zu arbeiten, muss eine Verbindung zur Datenbank aufgebaut werden. Dazu dient die Funktion odbcConnect():
Nach Enter können Sie die gewünschte Excel-Datei auswählen (Bild 3)...
Bild 3
...und sich ggf. die Verbindungsdaten anzeigen lassen:
Mit der Funktion sqlTables() können Sie sich die verwendeten Tabellen der Excel-Datei darstellen lassen:
Die Excel-Datei Test.xlsx enthält ein Tabellenblatt mit dem Namen Blatt1. Eine Übersicht über die Spalten der Tabelle Blatt1 erhalten Sie mit der Funktion sqlColumns(Verbindung, sqtable = "Blatt1"), die aber hier nicht ausgeführt wird (die Ausgabe ist sehr Umfangreich).
Den Tabelleninhalt von Blatt1 können Sie zur weiteren Bearbeitung in R mit der Funktion sqlFetch() als data.frame laden:
> Inhalt <- sqlFetch(Verbindung, sqtable = "Blatt1")
> str(Inhalt)
'data.frame': 9125 obs. of 26 variables:
$ Ereignis: num 0 0 1 1 0 0 0 1 1 1 ...
$ Einfluss: num 10.2 12.1 14.7 14.9 14.9 ...
$ Nr#1 : num 2 1 2 1 2 2 2 2 1 2 ...
$ Nr#2 : num 79 29 61 70 24 63 78 87 62 84 ...
$ Nr#3 : num 0 0 0 0 0 0 0 0 1 0 ...
$ Nr#4 : num 55 55 29 65 60 60 60 36 20 66 ...
$ Nr#5 : num 46.1 204.2 59.9 91 161.1 ...
$ Nr#6 : num 35.22 4.4 69.24 13.55 1.22 ...
$ Nr#7 : num 2 2 ....
|

|
|
Funktionsaufruf, ::-Argument
|
Das ::-Argument ist hilfreich, wenn Sie eine bestimmte Funktion aus einem Paket verwenden möchten, ohne das gesamt Paket in die R-Arbeitsumgebung zu laden. Das Paket wird nicht im Suchpfad der Arbeitsumgebung angezeigt. Das folgende Beispiel zeigt den Funktionsaufruf der Funktion kurtosis() aus dem Paket e0171 ohne das Paket in die Arbeitsumgebung zu laden:
|

|
|
Funktionsmodell, apply-Familie
|
Die apply-Funktionsfamilie beinhaltet Funktionen, die es ermöglichen, R-Funktionen wie z. B. mean() auf einem Datenobjekt wie z. B. einem Array, einer Matrix oder einem Datensatz anzuwenden, ohne das Schleifen-Kontrollstrukturen verwendet werden müssen. Dabei kann die R-Funktion auf das gesamte Daten-Objekt oder nur auf einem Teil angewendet werden. Das hört sich ein wenig komplex an, wird aber in den folgenden Beispielen anschaulich beschrieben. Natürlich wird die apply-Familie in der R-Literatur behandelt und dieser Beitrag dient nur als praktische Ergänzung!
- Die tapply-Funktion ermöglicht die elementweise Anwendung einer R-Funktion auf Beobachtungen, gesammelt in einem Datensatz (data.frame). Als Beispieldatensatz wird der airquality-Datensatz verwendet. Der Datensatz beinhaltet die Variablen Temp und Month. In diesem Beispiel werden wir die Durchschnittstemperatur pro erfassten Monat über die Funktion mean() schätzen:
> Daten <- airquality
> str(Daten)
'data.frame': 153 obs. of 6 variables:
$ Ozone : int 41 36 12 18 NA 28 23 19 8 NA ...
$ Solar.R: int 190 118 149 313 NA NA 299 99 19 194 ...
$ Wind : num 7.4 8 12.6 11.5 14.3 14.9 8.6 13.8 20.1 8.6 ...
$ Temp : int 67 72 74 62 56 66 65 59 61 69 ...
$ Month : int 5 5 5 5 5 5 5 5 5 5 ...
$ Day : int 1 2 3 4 5 6 7 8 9 10 ...
> MW_Temp <- tapply(Daten$Temp, Daten$Month, mean)
> MW_Temp
5 6 7 8 9
65.54839 79.10000 83.90323 83.96774 76.90000
|

|
|
Funktionsmodell, Aufruf
|
Ein z. B. lineare Modell wird über den Funktionsaufruf lm(y ~ x) geschätzt. Möchten Sie diesen Aufruf auf beispielsweise folgende Funktion
anwenden, muss die arithmetische Bedeutung im Modell berücksichtigt werden. Ein Aufruf in dieser Form
zeigt folgendes Ergebnis:
Obiges Ergebnis ist sicher nicht das gewünschte Ergebnis.
Durch die Funktion I()wird die arithmetische Bedeutung beibehalten.
|

|
|
Funktionsmodell, data.frame
|
Im obigen Beispiel wurde der Funktion lm() mit dem Aufruf jede unabhängige Variable explizit genannt. Liegen Ihre Daten als data.frame vor und sollen alle unabhängigen Variablen in das zu schätzende Modell einfließen, müssen Sie diese nicht einzeln eingeben, sondern können den .-Operator nutzen:
> Daten # Beispiel data.frame
y x1 x2 x3
1 1.0 0.5 0.03 0.20
2 2.1 1.2 0.07 0.45
3 3.1 1.5 0.13 0.60
4 3.9 2.1 0.22 0.80
5 5.0 2.6 0.30 1.00
6 5.8 3.0 0.32 1.20
7 7.3 3.6 0.41 1.50
8 8.1 4.1 0.46 1.76
9 9.2 4.6 0.55 1.90
10 10.3 5.5 0.62 2.10
# Nun kommt der .-Operator zum Einsatz:
> Modell <- lm(y ~., data = Daten)
> Modell
Call:
lm(formula = y ~ ., data = Daten)
Coefficients:
(Intercept) x1 x2 x3
0.2199 0.3829 3.8614 2.6589
Über den .-Operator werden bequem alle unabhängigen Variablen in das Modell eingebunden.
|

|
|
“Globale” Variable
|
Möchten Sie eine Variable (Datenstruktur) auch außerhalb einer Funktion dem Anwender zur Verfügung stellen, können Sie dies über das Zuweisungszeichen “<<-” erreichen. Hierbei sollten Sie aber die Eindeutigkeit um nicht in Konflikt mit anderen Objekten zu kommen bedenken. Empfehlen möchte ich Ihnen z. B. folgendes Konstrukt:
Funktion ist die Funktionsbezeichung und GlobaleVariable die Bezeichnung der globalen Variable. Diese Zusammensetzung der globalen Variable stellt eine halbwegs eindeutige Möglichkeit dar.
|

|
|
Grafik, Ausgabe als Datei
|
Möchten Sie die grafische Ausgabe nicht auf den Bildschirm darstellen, sondern als Datei ausgeben, verwenden Sie die Funktionen jpeg(), bmp() oder png():
Die Ausgabedatei befindet sich nun in dem zuvor eingestellten Verzeichnis:
|

|
|
Grafik, Ausgabe mehrere Grafiken in einer Darstellung
|
Die simple Frage “Wie können z. B. 2 Grafiken zusammen dargestellt werden?” kann wie folgt beantwortet werden:
> plot(sin,0,2*pi,ylab="")
> plot(cos,0,2*pi,ylab="",col= "red", )
Der Schlüssel ist hier das Argument add=TRUE!
|

|
|
Grafik, Ausgabe mehrere Darstellungen
|
Möchten Sie mehrere Grafiken in einem Grafikfenster ausgeben, können Sie dies über das Argument fig der der Funktion par() erreichen. Mit dem Argument fig werden die Ausgabebereiche des Grafikfensters angesprochen. Folgendes Beispiel soll dies verdeutlichen:
Das Objekt x beinhaltet die darzustellenden Daten. Über par() mit dem Argument fig wird festgelegt, dass unten 35% der Grafikfläche für den Boxplot zur Verfügung gestellt wird. Der Boxplot wird horizontal ausgegeben:
> x <- rnorm(100, 95,1)
> par(fig=c(0,1,0,0.35))
> boxplot(x, horizontal=TRUE)
Über die nächste par()-Funktion werden die restlichen 75% des Grafikfensters zur Ausgabe des Histogramms festgelegt. Das Histogramm wird als Dichtefunktion (prob)mit einer gestrichelten Linie (lines) dargestellt:
> par(fig=c(0,1,.25,1), new = TRUE)
> hist(x, prob=TRUE)
> lines(density(x), lty=2)
|

|
|
Grafik, Streudiagramm und Boxplot
|
Die Grafikfunktion boxplot() verfügt über das Attribut add und damit sind Sie in der Lage, ein Boxplot zur ergänzenden Darstellungen z. B. zu einem Streudiagramm zusätzlich anzuzeigen. Diese Möglichkeit ist eine Variante zu der hier beschriebenen. Beispiel:
> x <- rnorm(100, 10,0.5)
> plot(x)
> boxplot(x,range = 0, add = T, boxwex = 5, col = "grey", at = -1)
Über das Attribut add = T wird der Boxplot zur Grafik addiert, mit dem Attribut boxwex = 5 wird die “Breite” des Plots und über at = -1 wird die Position (ein bisschen ausserhalb der dargestellten Daten) festgelegt:
|

|
|
Grafik, Boxplot, verschiedene Möglichkeiten
|
Das Video Boxplot mit R beschreibt verschiedene Möglichkeiten der Boxplot-Darstellung in R. Und das nur nur mit R-Basis-Grafik-Möglichkeiten, also einfach und effektiv. Ein Appetitanreger:
|

|
|
Grafik, mehrere Histogramme darstellen
|
Möchten Sie mehrere Histogramme darstellen, hilft die Funktion multhist() des Paketes plotrix weiter:
> Daten <- list(rnorm(50, mean=3),rnorm(50,mean=5, sd=2),rnorm(50,mean=4))
> multhist(main="3 Histogramme", Daten)
|

|
|
Grafik, mehrere Histogramme darstellen , 2. Möglichkeit
|
Eine weitere Möglichkeit zur Darstellung von mehreren Histogrammen in einer Darstellung, zeigt folgendes Beispiel:
# Darstellung des 1. Histogramms:
> hist(Histogramm_1, col=rgb(0.1,0.1,0.1,0.5),xlim=c(0,10), ylim=c(0,250), main="Überlappende Histogramme", xlab="Wertebereich", ylab="Häufigkeit")
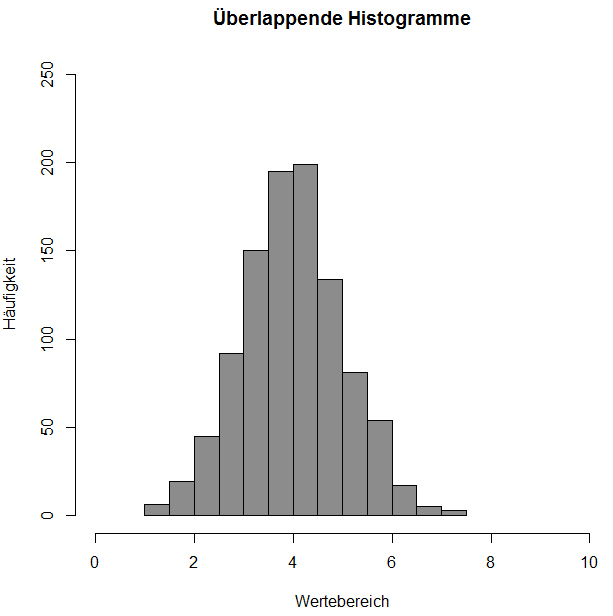
# Nun stellen wir das 2. Histogramm dar:
# Über das Funktionsargument add = True wird es zum bestehenden Histogramm addiert:
> hist(Histogramm_2, col=rgb(0.8,0.8,0.8,0.5), add = T)
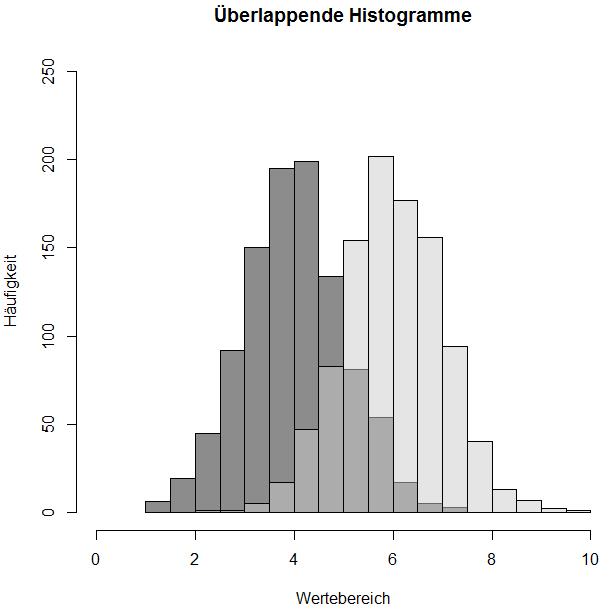
Der Überlappungsbereich wird in einem “mittleren” Grau dargestellt.
|

|
|
Grafik, Ausgabe Histogramm-Daten
|
Möchten Sie zusätzlich beispielhaft zu einem Histogramm die Anzahl der Beobachtungen in einer Klasse ausgeben, kann als “Anregung” Ihnen sicher folgendes Code-Schnipsel weiterhelfen (Siehe auch hier!):
> x <- rnorm(10000, 0, 1)
> hx <- hist(x, breaks=50, plot=FALSE)
> plot(hx, col=ifelse(abs(hx$breaks) < 1.96, 3, 2), main="Histogramm", xlab="Ihre z-normierte Daten", ylab="Häufigkeit")
> text(hx$mids, hx$counts+2, label=c(hx$counts), pos = 3)
Die Anzahl der Klasseninhalte wird über die Funktion text() ausgegeben. Dabei bestimmt der Parameter pos die Textausgabeposition. Experimentieren Sie mit diesem Parameter und schauen Sie auch in die Hilfe zur Funktion text().
|

|
|
Grafik, Bereiche unterschiedlich “einfärben”
|
Es kann hilfreich sein, z. B. in einem Histogramm Bereiche unterschiedlich farblich darzustellen. Als “Anregung” soll Ihnen folgendes Code-Schnipsel dienen:
> x <- rnorm(10000, 0, 1)
> hx <- hist(x, breaks=50, plot=FALSE)
> plot(hx, col=ifelse(abs(hx$breaks) < 1.96, 3, 2), main="Histogramm", xlab="Ihre z-normierte Daten", ylab="Häufigkeit")
Interessant ist hier die ifelse()-Funktionalität! Ist der Absolutwert kleiner als 1,96 wird der Farbwert 3 für grün (= yes) und wenn größer als 1,96 wird der Farbwert 2 für rot (= no) verwendet.
|

|
|
Grafik, Balkendiagramm, geordnet
|
Daten können anschaulich über ein Balkendiagramm visualisiert werden. Als Beispiel wird hier der Datensatz mtcars verwendet:
Die Variable mpg (Miles/(US) gallon) soll als Balkendiagramm dargestellt werden. Um eine übersichtliche Grafik zu erhalten, wird eine Tabelle mit selbst definierten Klassen erstellt:
> Tabelle <- table(cut(mtcars$mpg, breaks = 10))
> Tabelle
(10.4,12.8] (12.8,15.1] (15.1,17.5] (17.5,19.8] (19.8,22.1] (22.1,24.5] (24.5,26.9] (26.9,29.2] (29.2,31.6] (31.6,33.9]
2 4 6 6 5 3 1 1 2 2
Auf Basis dieser Tabelle wird in den meisten Fällen vom Datenanalyst folgende Grafik erzeugt:
> barplot(Tabelle, main = "Verbrauch in Meilen pro Gallone", xlab = "Verbrauchklasse", ylab = "Anzahl Fahrzeuge", ylim = c(0,7))
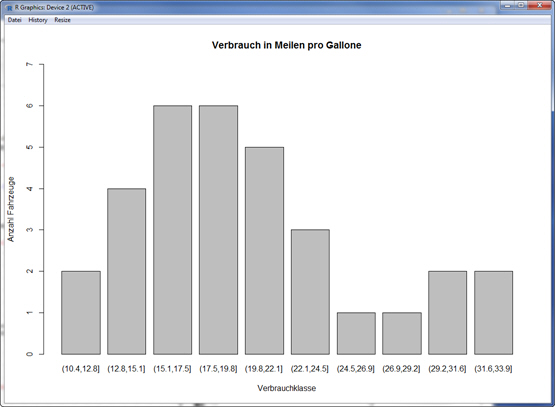
Möchten Sie aus Ihnen bekannten Gründen die Klassen nach fallenden Häufigkeiten von links nach rechts sortieren, lässt sich das wie folgt erreichen:
> Tabelle <- sort(Tabelle, decreasing = TRUE)
> barplot(Tabelle, main = "Verbrauch in Meilen pro Gallone", xlab = "Verbrauchklasse", ylab = "Anzahl Fahrzeuge", ylim = c(0,7))
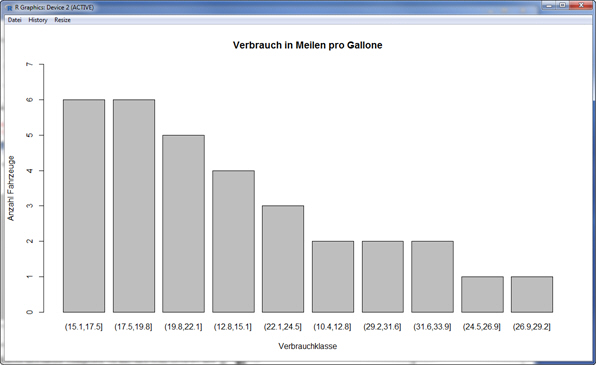
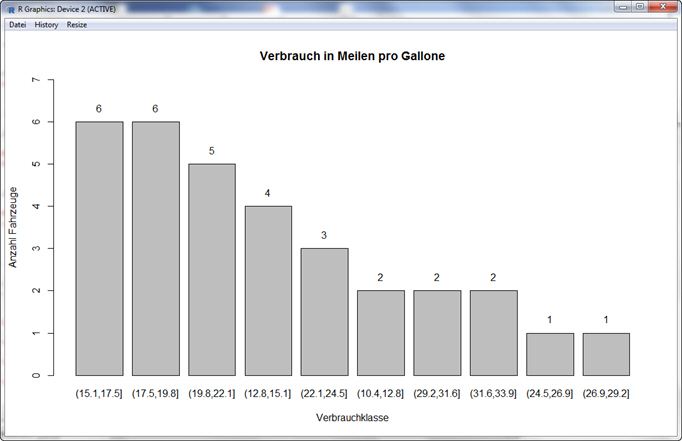
Die obige Darstellung lässt sich noch ausbauen. Möchten Sie noch die Häufigkeit (Anzahl der Fahrzeuge) über den Balken darstellen (Label), lässt sich dies wie folgt erreichen:
> # Label erstellen, wobei die Labels die Häufigkeit darstellen. Diese können wir
ebenfalls als y-Position nutzen.
> Label <- c(6,6,5,4,3,2,2,2,1,1)
> # Um die x-Achsen-Koordinaten zu erhalten, wird der barplot in Diagramm abgelegt:
> Diagramm <- barplot(Tabelle, main = "Verbrauch in Meilen pro Gallone", xlab = "Verbrauchklasse", ylab = "Anzahl Fahrzeuge", ylim = c(0,7))
> # Nun werden die labels über die text-Funktion hinzugefügt:
> text(Diagramm[,1], Label + 0.1, labels = Label, pos = 3)
|

|
|
Grafik, Bereich unter einem Polygonzug einfärben
|
Besteht der Wunsch, die Fläche unter z. B. einem Polygonzug einzufärben, können Sie dieses über die Funktion polygon() erreichen:
> x <- 1:50
> y <- rnorm(50)
> plot(x,y)
> lines(x,y)
> polygon(cbind(c(min(x),x,max(x)),c(min(y),y,min(y))), col="grey")
Hier das Resultat:
|

|
|
Grafik, Farben
|
Möchten z. B. Grafiken mit unterschiedlichen Farben erzeugen, stellen Sie sich vielleicht die Frage, welche vordefinierte Farben R Ihnen zur Verfügung stellen kann! Hier hilft Ihnen die Funktion colors() weiter. Über die Konsole eingegeben gibt Sie Ihnen zur R-Version 2.10.1 1965 Farben aus.
Möchten Sie Ihre eigene Farbe kreieren, können Sie die über die Funktion rgb() erledigen:
> Farbe <- rgb(43,33,235, alpha = 200, maxColorValue = 255)
Die ersten 3 Werte beschreiben den Farbton, hier ein blau, und der alpha-Wert gibt die Helligkeit wieder. Als Beispiel werden ein paar Punkte grafisch ausgegeben:
> plot(1:6, pch = 20, col = Farbe)
|

|
|
Funktion auswerten und darstellen
|
Dieser Hinweis zeigt, wie Sie eine Funktion grafisch über R darstellen lassen können:
Im ersten Schritt erzeugen Sie eine Zeichenkette, die neben der darzustellenden Funktion die R-Funktion curve zur grafischen Ausgabe beinhaltet:
> FunkGraph = "curve(sin(cos(x)*exp(-x/2)),from = -8, to = 7, n = 2001, main = 'Funktionsgraph')"
# Ihre Funktion: sin(cos(x)*exp(-x/2))
# Parameter zur Funktion curve: from = -8, to = 7, n = 2001, main = 'Funktionsgraph'
# Achten Sie auf ‘-Zeichen für den Grafiktitel!
Im zweiten Schritt wird die Zeichenkette FunkGraph über die Funktion parse aufgelöst/analysiert und über eval ausgewertet:
Hier das Resultat:
|

|
|
Grafik, “Errorbar”, Abweichungen darstellen
|
Oft besteht der Wunsch, neben der Ausgabe einer z. B. linearen Funktion auch die Residuen grafisch darzustellen. Ohne Aufwand können Sie dies mit der Funktion errbar() aus dem Paket Hmisc erreichen. Ausgehend von folgendem Beispieldatensatz...
... wird Ihnen über den Funktionsaufruf...
> errbar(Beispiel$X, Beispiel$Y, Beispiel$Ymax, Beispiel$Ymin, xlab="X-Daten", ylab="Y-Daten", main="Residuen-Beispiel")
... neben den X-Y-Werten die Abweichungen (Ymin und Ymax) grafisch ausgegeben:
|

|
|
Grafik, Vertrauensbereich darstellen
|
Besteht die Notwendigkeit, den Mittelwert inklusive Angaben zur Streuung, wie z. B. die Standardabweichung, grafisch darzustellen, hilft die Funktion plotCI() des Paketes plotrix weiter. Das folgende kleine Skript zeigt die Darstellung des Mittelwertes inklusive des Vertrauensbereichs. Das Skript und dessen Ausführung können Sie sich auch unter Vertrauensbereich mit R als Video anschauen.
Gehen wir das Skript durch:
Im den ersten Schritten wird das Paket geladen und ein paar Zufallsdaten erzeugt:
## Lade benötigte Pakete:
library(plotrix) # Grafische Darstellung
## Datensimulation normalverteilter Beobachtungen:
mu_Merkmal <- 5 # Erwartungswert mu für das Merkmal
Merkmal <- rnorm(100, mu_Merkmal, 1) # n = 100, Mittelwert = mu_A, Standardabw. = 1
Danach werden Parameter der simulierten Normalverteilung geschätzt, weil die Standardabweichung Stabw und der Freiheitsgrad df für die Vertrauensbereichschätzung VB benötigt wird:
## Schätzen der Parameter Mittwelwert und Standardabweichung der Beobachtungen:
MW <- mean(Merkmal); MW # Mittelwert
Stabw <- sd(Merkmal); Stabw # Standardabweichung
n <- length((Merkmal)); n # Anzahl Beobachtungen (es ist klar, es sind 100!)
df <- n - 1; df # Freiheitsgrad
In den nächsten Schritten werden die Parameter zur Berechnung des Vertrauensbereichs VB berechnet...
P <- 1 - 0.05/2 # Wahrscheinlichkeit P = 95%, zweiseitig, zur Berechnung des
T-Verteilungsquantils
t_Quant <- qt(P, df) # Quantil der t-Verteilung
... und über die Formel
der Vertrauensbereich VB berechnet:
VB <- t_Quant *Â Stabw / sqrt(n); VB # Berechnung des Vertrauensbereiches
VB_unten <- MW - VB; VB_unten # untere Vertrauensbereichgrenze
VB_oben <- MW + VB; VB_oben # obere Vertauensbereichgrenze
Wir kennen nun für diese zufällig erzeugten Daten den Mittelwert MW und den Vertrauensbereich VB und können ihn über die Funktion plotCI grafisch darstellen. Das erste Argument ist der Mittelwert MW und das zweite Argument uiw nimmt den Vertrauensbereich VB auf und übergeben sie an die Funktion plotCI:
## Grafische Darstellung des Vertrauensbereiches:
plotCI(MW, uiw = VB, lwd = 2, col="blue", scol = "red",
main = "Mittelwert inklusive Vertrauensbereich",
xaxt = "n", xlab = "Merkmal", ylab = "Werte der Simulation")
Der Vertrauensbereich beinhaltet mit der gewählten statistischen Wahrscheinlichkeit P den Erwartungswert µ der simulierten Normalverteilung. Um das grafisch zu verdeutlichen, zeichnen wir unser geheimes Wissen, den Erwartungswert µ als Strich in die Grafik ein:
## Darstellen des Erwartungswertes als Linie:
abline(h = mu_Merkmal, lty = 2)
text(1.1, mu_Merkmal+0.015, "Erwartungswert")
Führen Sie das Skript aus, sollten Sie eine grafische Darstellung ähnlich dieser Darstellung erhalten.
Auf die Abbildung der geschätzten Parameter wurde im obigen Skript verzichtet, da sie von den zufällig erzeugten Beobachtungen abhängen.
|

|
|
Grafik, Interaktive- networkD3
|
Über das D3-JavaScript-Network können ansehnliche interaktive Grafiken als html-Dateien erzeugt werden. Ein Beispiel dazu:
Über das Paket networkD3 lassen sich interaktive Netzwerkgrafiken auf Basis des htmlwidgets-Rahmen erzeugen. Hier ein Beispiel dazu:
> library(magrittr) # Brauchen wir später zum Speichern
> library(networkD3)
> src <- c("A", "A", "A", "A","B", "B", "C", "C", "D")
> target <- c("B", "C", "D", "J", "E", "F", "G", "H", "I")
> networkData <- data.frame(src, target)
> networkData
src target
1 A B
2 A C
3 A D
4 A J
5 B E
6 B F
7 C G
8 C H
9 D I
Über die Funktion simpleNetwork() wird eine HTML-Datei im htmlwidgets-Framework erzeugt und als interaktive Grafik im Browser ausgegeben:
Mithilfe des magritter-Paketes, wird die Grafik gespeichert und lässt sich zu z. B. in eine HTML-Umgebung einbinden:
Beispiel_files enthält die nötigen Hilfsdateien. Mit einem Doppelklick auf die Datei Beispiel.html wird die Grafik im Browser angezeigt und "aktiv" angeboten.
|

|
|
Grafik, Legende (Achsenbeschriftun g)
|
Grafikfunktionen wie plot() nehmen Ihnen in der grafischen Darstellung Ihrer Daten schon viel Arbeit ab, indem Sie sich keine Gedanken über die Achsenformatierung und -beschriftung machen müssen. Möchten Sie irgendwann aber Ihre eigene Achsenbeschriftung vornehmen, ist diese Automatismus störend.
In dem hier beispielhaft verwendeten Datensatz würde in der grafischen Darstellung über plot() der Index als x-Achsenbeschriftung dargestellt. Als Beschriftung soll aber der zeitliche Verlauf der Zeitreihe als x-Achse ausgegeben werden, wie in der Grafik gelb markiert.
Dies kann in zwei Schritten erreicht werden. Im 1. Schritt wird die durch plot vorgegebene x-Achsenbeschriftung über den Parameter xaxt="n" ausgeschaltet:
> plot(Rohoel$Preis, type="o", main="Rohölpreis", ylab="Preisindex", xlab="Zeitraum", xaxt="n")
Das x-Achsenlabel Zeitraum wurde aus Platzgründen in obiger Grafik nicht dargestellt. Im 2. Schritt wird über die Funktion axis() die 98 Labelwerte aus dem Vektor Rohoel$Monat der x-Achse (1) zugeordnet und dargestellt:
> axis(1,at=seq(1:98), labels=Rohoel$Monat)
Die von Ihnen gewünschte Achse wird über den ersten Parameter, hier 1 für die x-Achse, der Funktion axis() angesprochen.
|

|
|
Grafik, Legende (Achsenbeschriftun g, Schriftyp)
|
Möchten Sie einen Teil der Legende besonders hervorheben, können Sie dieses wie im Folgenden Beispiel gezeigt erreichen:
> plot(Daten$Y ~ Daten$X, xlab=(expression(Das ~ sind ~ italic(meine) ~ x-Daten)), ylab=expression(Das ~ sind ~ bold(meine) ~ y-Daten))
(bold = fett, italic = kursiv)
Müssen Sie griechische Buchstaben im Legendentext darstellen, können Sie dieses über die Funktion expression() erreichen:
> Legende <- expression(paste(phi, " /", "cm"))
> plot(Daten, xlab <- Legende)
|

|
|
Grafik mit Lattice
|
Sollen Daten in Panel-Form dargestellt werden, hilft das lattice-Paket weiter. Ein Beispiel dazu:
Die Wachstumsrate (Growth.rate) soll nun über die Gruppierungsvariable Daphnia als Boxplot dargestellt werden:
bwplot(~ Growth.rate | Daphnia, data = Daten,
main = "Übersicht Wachstumsrate Daphnia",
xlab = "Wachstumsrate")
Das sieht schon recht gut aus und das Paket bietet eine Fülle von grafischen Möglichkeiten. Soll aber neben den angebotenen Standardparametern die Grafik angepasst werden, ist das mit ein wenig Aufwand verbunden. Im folgenden Beispiel, wird der “orange” Hintergrund strips nach hellblau geändert:
strip.backround <- trellis.par.get("strip.backround")
strip.backround$col <- "lightblue"
trellis.par.set("strip.background", strip.backround)
bwplot(~ Growth.rate | Daphnia, data = Daten,
main = "Übersicht Wachstumsrate Daphnia",
xlab = "Wachstumsrate")
Über trellis.par.get und trellis.par.set können die Grafikparameter angepasst werden, ist aber - dass muss ich zugeben - mit Aufwand verbunden.
|

|
|
Grafik, Legende
(Zahlendarstellung)
|
Möchten Sie z. B. die Daten der X-Achse z. B. mit einem Tausender-Trennzeichen versehen, können Sie dieses über die Funktion prettyNum() erreichen. Folgendes Beispiel zeigt die Funktionsweise:
Die x-Daten sollen die zukünftige X-Achse darstellen:
Die x-Daten werden zu Label-Daten über prettyNum() aufgehübscht:
> x_label <- prettyNum(x, big.mark = ".")
> x_label
[1] "10.000" "20.000" "30.000" "40.000"
Die y-Daten werden grafisch ausgegeben und dabei wird wie im obigen Beispiel die X-Achse zuerst unterdrückt...
> plot(y, type="o", main="prettyNum-Beispiel", ylab="Y-Achse", xlab="X-Achse", xaxt="n")
... und nun als X-Achse die Labels ausgegeben:
> axis(1, at=seq(1:4), labels=x_label)
Hier das Resultat:
|

|
|
Hilfe, zusätzliche Unterstützung
|
Über das Paket SOS erhalten zusätzliche nützliche Hilfefunktionen. Nach dem Laden des Paketes erhalten Sie über die ???-Funktion Informationen in welchen Paketen (wenn möglich) die gesuchte Funktion enthalten ist. Suchen Sie z. B. die spline-Funktion gehen Sie wie folgt vor:
Das Suchergebnis wird in dem Objekt Ergebnis abgelegt und die Anzahl der Treffer ausgegeben:
Rufen Sie das Ergebnis auf ...
... wird Ihnen das Suchergebnis im Browser angezeigt:
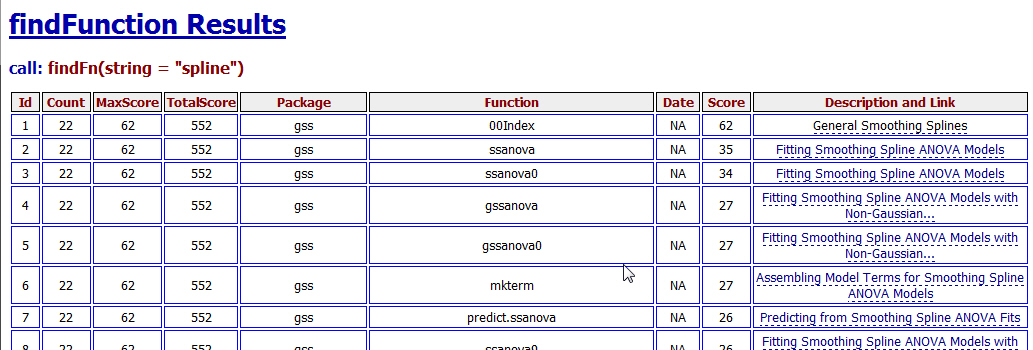
Klicken Sie im linken Bereich Description and Link auf einen entsprechenden Link, wird Ihnen die HTML-Hilfe zum Paket bzw. zur Funktion angezeigt!
|

|
|
Hilfe, Tab-Taste
|
Sie kennen die Hilfe-Funktion apropos() über der Sie, wenn Sie nur Bruchstücke einer Funktion kennen, eine Auflistung der möglichen Funktionen auf der Konsole ausgegeben bekommen.
Eine interessante Möglichkeit ist die Tab-Tasten-Funktion. Das folgende Beispiel geht davon aus, dass Sie nur die ersten beiden Buchstaben “me” der gesuchten Funktion kennen. Um die möglicherweise passenden Funktionen angezeigt zu bekommen, geben Sie me auf der Konsole ein und drücken dann die Tab-Taste. Ihnen werden dann die Funktionen angezeigt, die mit me anfangen:
> me
mean mean.data.frame mean.Date
mean.default mean.difftime mean.POSIXct
mean.POSIXlt median median.default
medpolish mem.limits memCompress
memDecompress memory.limit memory.profile
memory.size menu merge
merge.data.frame merge.default mergeMethods
message metaNameUndo method.skeleton
methods methodSignatureMatrix methodsPackageMetaName
methods::
> me
Entscheiden Sie sich beispielsweise für die Funktion mean, geben Sie einfach die fehlenden Zeichen an ein. Mit der Zeicheneingabe wird die Ausgabe der möglichen Funktion automatisch gelöscht! Sie können auch die gewünschte Funktion mit der Maus markieren, kopieren und dann hinter dem Eingabeprompt einfügen. Über die weitere Zeicheneingabe in Verbindung mit der Tab-Taste können Sie das “Angebot” der möglichen Funktionen verfeinern.
|

|
|
if-Variante
|
Im Folgenden wird eine verkürzte if-Abfrage beschrieben. Aber Vorsicht! Denken Sie hier bei der Verwendung an die Lesbarkeit des Codes!
Die Variable Daten beinhaltet z. B.: "a" "b" "b" "b" "c". Daten soll auf b’s geprüft werden und wenn vorhanden, gegen z’s ausgetauscht werden:
|

|
|
Interpolation
|
Eine einfache aber dennoch oft gestellte Frage: Wie werden in R Zwischenwerte interpoliert? Sie erreichen dies über die Funktion approx(). Im Folgenden Beispiel werden die Werte zwischen 1 und 10 linear interpoliert:
> x <- c(0, 10)
> resultat <- approx(x, method="linear")
> resultat$y
[1] 0.0000000 0.2040816 0.4081633 0.6122449 0.8163265 1.0204082
[7] 1.2244898 1.4285714 1.6326531 1.8367347 2.0408163 2.2448980
[13] 2.4489796 2.6530612 2.8571429 3.0612245 3.2653061 3.4693878
[19] 3.6734694 3.8775510 4.0816327 4.2857143 4.4897959 4.6938776
[25] 4.8979592 5.1020408 5.3061224 5.5102041 5.7142857 5.9183673
[31] 6.1224490 6.3265306 6.5306122 6.7346939 6.9387755 7.1428571
[37] 7.3469388 7.5510204 7.7551020 7.9591837 8.1632653 8.3673469
[43] 8.5714286 8.7755102 8.9795918 9.1836735 9.3877551 9.5918367
[49] 9.7959184 10.0000000
Durch den Vorgabewert n = 50 werden 50 Zwischenwerte interpoliert. Die Hilfe zur Funktion approx() zeigt weitere Möglichkeiten.
|

|
|
Menü,
Funktionsintern
|
Möchten Sie in einer Funktion dem Anwender ein Auswahlmenü anbieten, hilft hier die Funktion menu() weiter:
Auswahl <- c(menue_text_1, menue_text_2)
switch(menu(Auswahl, title = "Menübeispiel"), Auswahl <- 1, Auswahl <- 2)
In einer Funktion eingebunden, könnte die Auswahl wie folgt aussehen:
Obige Beispielfunktion können Sie hier downloaden.
|

|
|
Menü,
als Bestandteil des GUI-Menüs
|
Möchten Sie das GUI-Menü der R-Konsole mit einem eigenen Menü erweitern, können Sie dies über die Funktionen windMenuAdd() bzw. winMenuAddItem() erreichen:
Über die Funktion winMenuAddItem() wurde die Menü-Überschrift Zusatz Menü, der Menü-Punkt Starte Grafik der mit der Funktion run.GrapheR() verknüpft ist, zugefügt:
Das Ganze lässt sich noch so verfeinern, dass das Zusatz-Menü direkt mit dem Starten der R-GUI eingebaut wird (siehe hier)!
Das hinzugefügte Menü lässt sich über die Funktion winMenuDel() wieder entfernen:
|

|
|
PDF-Datei
|
Möchten Sie eine grafische Ausgabe als PDF-Datei speichern, können Sie die grafische Ausgabe über die pdf()-Funktion als PDF-Datei in das eingestellte Verzeichnis umleiten:
> pdf(file="Test.pdf")
> hist(Daten, main="Beispielhistogramm")
> dev.off()
Das Objekt Daten beinhaltet die Beispieldaten und ist nicht weiter spannend. Hier geht es nur darum, die Ausgabe des Histogramms umzuleiten, Die offene PDF-Datei, die hier die Bezeichnung Test.pdf trägt, wird durch die Funktion dev.off() geschlossen. Danach kann die PDF-Datei Acrobat geöffnet werden:
|

|
|
Programmier-Tipp:
Funktionsaufruf mit GUI-Start
|
Möchten Sie mit dem Start der R-GUI, der R-Konsole, direkt eine Funktion in die Arbeitsumgebung laden, können Sie das über die Datei Rprofile.site erreichen:
In diesem Beispiel soll ein Zusatz-Menü dem R-GUI-Menü hinzugefügt werden. Das Menü soll den Anwender in die Lage versetzten, über das Menü Start Grafik die Funktion run.GrapheR() aufrufen. Dazu wird der Funktion .First die entsprechend ausgeprägten Funktion winMenuAddItem() übergeben (nächste Abbildung). Vorher wird das Paket GrapheR über die Funktion library() in die Arbeitsumgebung geladen.
Mit dem Start der R-GUI wird die Datei Rprofile.site durchlaufen und das Zusatz-Menü Zusatz Grafik installiert und angeboten:
Das Laden des Paketes GrapheR und des notwenigen Paketes tcltk wird in der GUI als Info ausgegeben.
|

|
|
Programmier-Tipp:
Ausnahmen behandeln
|
Sie entwickeln eine Funktion, die eine vom Nutzer anzugebende Datei öffnen und verarbeiten soll. Dabei kann es passieren, dass der Nutzer sich verschreibt. Der Dateiname oder das Verzeichnis entspricht nicht der Erwartung, somit lässt die Datei sich nicht öffnen und die Ausführung der Funktion wird mit einer entsprechenden Meldung abgebrochen.
Das Abbruchverhalten können Sie über die Behandlung von Ausnahmen steuern! Die folgende kleine Beispielfunktion Test soll eine csv-Datei einlesen und den Dateiinhalt auf der Konsole ausgeben:
> Datei
[1] "C:/Users/Günter/Documents/Statistik/Zwischen/Wirkstoff.csv"
> Test <- function(Datei) {
+ if(file.exists(Datei)) {
+ Daten <- read.csv2(Datei)
+ print(Daten)
+ } else {
+ stop("Datei kann nicht geöffnet werden! Datei: ", Datei)
+ }
+ }
Über die Funktion file.exists() wird auf das Vorhandensein der Datei geprüft. Ist die Datei nicht vorhanden, wird die stop()-Funktion im else-Teil ausgeführt. Die stop()-Funktion bricht die Abarbeitung der Funktion Test ab. Ist dieses Verhalten nicht nötig, kann die stop()-Funktion durch die warning()-Funktion ersetzt werden. In diesem Falle, wird die Funktion durchlaufen und zum Schluss einen Warnmeldung ausgegeben.
Zuerst soll das normale Verhalten - der Nutzer hat alles richtig eingegeben - gezeigt werden:
> Test(Datei)
Wirkstoff.A Wirkstoff.B Wirkstoff.C Pruefer Datum
1 99.1 97.3 95.3 Karl 10.08.10
2 99.3 97.5 95.1 Karl 11.08.10
3 98.8 96.9 95.6 Peter 12.08.10
4 98.9 97.2 95.7 Karl 13.08.10
5 99.3 97.4 95.1 Peter 14.08.10
6 99.2 97.1 95.5 Peter 15.08.10
7 99.5 97.0 95.6 Karl 16.08.10
8 99.4 96.9 95.4 Peter 17.08.10
9 99.0 97.4 95.8 Peter 18.08.10
Und nun das Verhalten, wenn die Datei Wirkstoff.csv nicht in dem angegebenen Verzeichnis vorhanden ist:
> Test(Datei)
Fehler in Test(Datei) :
Datei kann nicht geöffnet werden! Datei: C:/Users/Günter/Documents/Statistik/Zwischen/Wirkstoff.csv
|

|
|
Programmier-Tipp:
Name des übergebenen Datensatzes
|
Möchten Sie die Bezeichnung des an einer programmierten Funktion übergebenen Datensatzes auslesen und z. B. in der Funktionsausgabe ausgeben, hilft die Funktionkombination deparse(substitute(x)) weiter:
Die Ausgabe kann dann wie folgt aussehen:
> Untertitel <- paste(Untertitel, " (Datensatz: ", Fakt.Daten.Namen, ")")
Das Beispiel wurde in der Funktion Faktorenanalyse realisiert. Der übergebene Datensatz trägt die Bezeichnung Fakt.Beispiel:
> faktorenanalyse(Fakt.Beispiel,labelsp=T,info=T,grafik=T)
Die grafische Ausgabe zeigt dann die Bezeichnung des Datensatzes im Untertitel:
|

|
|
Programmier-Tipp: Nutzer-Eingabe abfragen
|
Möchten Sie dem Nutzer während der Funktionsausführung Gelegenheit geben, den Funktionsablauf zu beeinflussen oder Eingaben durchzuführen, bietet R Ihnen die Funktion winDialog() an.
Möchten Sie dem Anwender Möglichkeiten zur Beeinflussung des Programmablaufes geben, können Sie die Funktion winDialog() wie folgt nutzen:
> Nachricht <- "Meine Nachricht!" #Nachricht für den Nutzer
> # In dem Objekt Eingabe wird die Eingabe abgelegt:
> Eingabe <- winDialog(type = c("yesnocancel"),Nachricht)
Durch obige Funktion wird dem Nutzer folgendes Dialog-Fenster dargestellt:
Wie Sie erkennen können, bestimmt das Argument type die angebotenen Schaltflächen. Es stehen Ihnen die Möglichkeiten “ok”, “okcancel”, “yesno” und “yesnocancel” zur Verfügung. Die durch den Nutzer gedrückte Schaltfläche - das Ereignis - wird in der Variablen Eingabe abgelegt:
Im hier dargestellten Beispiel wurde die Schaltfläche Ja gedrückt und damit “YES” als Zeichenkette in der Variable Eingabe abgelegt. Die Variable lässt sich nun prüfen und z. B. dementsprechend eine Programmverzweigung durchführen.
Es können vom Nutzer auch konkrete Eingaben über die Funktion winDialogString() abgefragt werden. Im Folgenden soll der Nutzer eine Eingabe in dem Wertebereich von 0 bis 10 durchführen, wobei 1,5 als Vorgabewert festgelegt wird:
> Nachricht <- "Bitte geben Sie den Wertebereich von 0 bis 10 ein:"
> Vorgabe <- "1.5"
> Eingabe <- winDialogString(Nachricht, Vorgabe)
Der Funktionsaufruf zeigt dann folgendes Dialog-Fenster:
Geben Sie z. B. 2,3 ein, ist der Inhalt der Variablen Eingabe gleich “2.3”. Möchten Sie die Eingabe tatsächlich in einer weiteren Berechnung verwenden, denken Sie an die Typwandlung:
|

|
|
Programmier-Tipp:
Fortschrittsbalken
|
Umfangreichere Berechnungen (Schätzungen) benötigen entsprechende Systemkapazitäten Ihres Computers. Möchten Sie nicht im Unklaren über den Status sein, können Sie sich über einen Fortschrittsbalken über den Berechnungs-Fortschritt machen:
Sie kennen derartige Funktion aus diversen Programmen und möchten Sie diese Funktionalität in Ihren R-Funktionen nutzen, laden Sie hier die Beispielfunktion auf Ihren Computer runter!
|

|
|
Programmier-Tipp:
beliebiger Programmaufruf / URL Aufruf
|
Besteht die Notwendigkeit aus einer Funktion z. B. ein Windows-Programm aufzurufen, können Sie dies über die shell.exec()-Funktion erreichen. Im Folgendem Beispiel wird der Eitor Ultraedit aufgerufen:
> shell.exec("C:/Program Files/IDM Computer Solutions/UltraEdit-32/uedit32.exe")
Über diese Funktion lassen sich auch URL’s aufrufen:
> shell.exec("http://www.faes.de")
Das URL-Beispiel können Sie direkt ohne Anpassungen ausführen!
|

|
|
OpenOffice
|
Mit dem Paket odfWeave können Sie Ihre Daten oder Grafiken immer aktuell in einem OpenOffice-Dokument einbinden. D. h., Sie können aus der R-Umgebung Informationen an ein OpenOffice-Textdokument schicken und verfügen dadurch immer über einen aktuellen Bericht. Dazu sind einige wenige Schritte erforderlich.
Im 1. Schritt muss das Paket odfWeave installiert werden. Nutzen Sie die R-Version 2.5.1 sollten folgende Pakete geladen sein:
Im obigen Beispiel wurden die Pakete XML und lattice vom Paket odfWeave nachgeladen. Um nun im 2. Schritt z. B. den Datensatz Daten in einem OpenOffice-Textdokument auszugeben, sind folgende Anweisungen zwischen << und dem @ im Textdokument nötig:
Dieses OpenOffice-Textdokument speichern Sie z. B. unter den Namen Test.odt und als 3. Schritt geben Sie auf der R-Konsole folgende Anweisung ein:
odfWeave("C:/Programme/Statistik/R-2.5.1/Eigene_Projekte/Test.odt",
"C:/Programme/Statistik/R-2.5.1/Eigene_Projekte/Test2.odt")
Mit dieser Anweisung müssen Sie auch die Zieldatei Test2.odt angeben! Die odfWeave-Funktionalität erstellt eine neue Datei, die Zieldatei, um dort den Inhalt von Test.odt und die R-Informationen abzuspeichern. So könnte die Zieldatei Test2.odt aussehen:
Auch Grafiken lassen sich einbinden. Das nächste Bild zeigt das Einbinden der plot()-Funktion ...
... und nach dem Aufruf der obigen R-Anweisung wird Test2.odt erneut erzeugt und zeigt nun zusätzlichen Inhalt:
Hier können Sie die Datei Test.odt zur Ansicht laden. Das Paket odfWeave bietet noch weiter Möglichkeiten, schauen Sie einfach mal...!
|

|
|
R im Batch-Modus
|
Möchten Sie R ohne Benutzeroberfläche ausführen, bietet sich der R-Batch-Modus an. Im Folgenden wird ein kleines Beispiel Schritt-für-Schritt für das OS Windows gezeigt:
- Windows-Umgebungsvariablen
Um den Batchmodus R-Verzeichnisunabhängig aufrufen zu können, stellen Sie sicher, dass die Betriebssystem-Umgebungsvariable PATH so eingestellt ist, dass R.exe gefunden werden kann (Bild 1 & 2):
 Bild 1 Bild 1
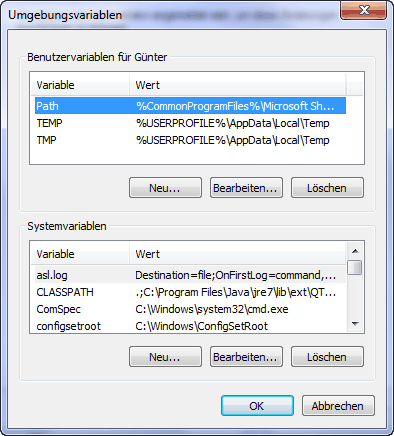 Bild 2 Bild 2
Abhängig von Ihrem System sollte ein ähnlicher Eintrag vorhanden sein: c:\Program Files\R\R-3.0.1\bin
- Die R-Skript-Datei
Die R-Funktionen, die Sie im Batch-Modus ausführen möchten, legen Sie in einem Editor Ihrer Wahl fest (Bild 3) ...
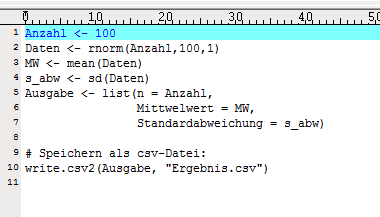 Bild 3 Bild 3
... und speichern die Datei, hier beispielhaft Batchskript.txt genannt in einem Verzeichnis ebenfalls Ihrer Wahl (Bild 4):
 Bild 4 Bild 4
- Die Windows-Batch-Datei
In der Windows-Batch-Datei legen Sie fest, in welchem Verzeichnis sich die Batchskript.txt-Datei befindet und wo die Ausgabe gespeichert werden soll (Bild 5, Zeile 6). Wesentlich ist natürlich der R-Aufruf mit den Eingabe- und Ausgabe-Dateien (Bild 5, Zeile 8):
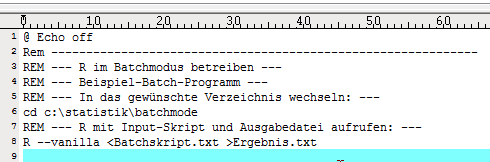 Bild 5 Bild 5
Diese Batch-Datei speichern Sie in einem beliebigen Verzeichnis, hier als R-Batch.BAT (Die Endung BAT ist wichtig zur Ausführung über cmd.exe!) und in diesem Beispiel wird eine Verknüpfung auf dem Desktop abgelegt (Bild 6):
 Bild 6 Bild 6
Zur Erinnerung, das Verzeichnis Batchmode (Bild 4) beinhaltet im Augenblick nur die Datei Batchskript.txt!
- Die Ausführung der Batch-Datei R-Batch.BAT
Mit einem Doppelklick auf das Symbol (Icon) R-Batch.BAT (Bild 6), wird in das angegebene Verzeichnis c:\statistik\batchmode gewechselt, R im stillen Modus aufgerufen (-- vanilla), dabei die Batchskript.txt-Datei eingelesen (< Symbol) und die Datei Ergebnis.txt (> Symbol) erzeugt. Der R -Prozess wird danach beendet.
- Das Resultat
Das R-Skript (Bild 3) enthält die Anweisung, eine Ergebnis.csv-Datei zu schreiben und in der Windows-Batch-Datei haben wir ebenfalls explizit eine Ergebnis.txt-Datei angegeben (wenn Sie das nicht machen, erzeugt R eine Datei). Das Verzeichnis Batchmode sollte bei Dateien beinhalten, was durch eine Prüfung bestätigt wird (Bild 7):
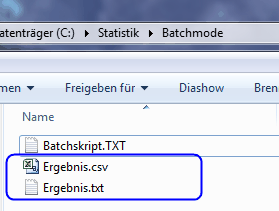 Bild 7 Bild 7
Der Inhalt der Ergebnis.txt-Datei hat ein wenig Log-Buch-Charakter und sieht wie folgt aus (Bild 8):
 Bild 8 Bild 8
Und die Ergebnis.csv-Datei öffnen wir mit MS Excel (Bild 9):
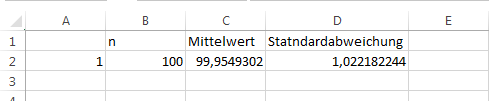 Bild 10 Bild 10
Fertig! Das obige Beispiel lässt sich natürlich noch deutlich ausbauen!
Von Zeit zu Zeit wird die Frage diskutiert, wie einem Batch-Aufruf Parameter für das aufzurufende R-Script mitgegeben werden können. Wird im Internet nach Möglichkeiten gesucht, finden sich einige Hinweis, die allerdings auch vom verwendeten Betriebssystem abhängen. Meines Erachtens, bietet sich an, eine eigene Parameter-Datei zur Parameterübergabe durch das R-Script einzulesen. Diese Parameter-Datei kann jederzeit überarbeitet werden, ohne dass die Batch-Datei oder das R-Script angefasst werden muss.
Ausgehend vom obigen Beispiel kann die Parameter-Datei wie folgt aussehen (als csv-Datei, Bild 11):
Bild 11
Die R-Script-Datei muss dann natürlich angepasst werden (Bild 12, gelbe Markierung):
Bild 12
Dieser Hinweis ist natürlich nur ein Denkanstoß!
|

|
|
R im Batch-Modus zeitgesteuert ausführen
|
Im Hinweis “R im Batch-Modus” wurde bereits gezeigt, wie man mittels einer Batch Datei ein R-Programm auch ohne gestartete R-Konsole ausführen kann. Diese Batch-Datei wird normalerweise mit einem Doppelklick oder Shell-Aufruf gestartet. Hier wird nun beispielhaft für das Betriebsystem Windows 7, 64 Bit gezeigt, wie man die Batch-Datei automatisiert vom Betriebssystem zu einem bestimmten Zeitpunkt starten lassen kann.
|

|
Rufen Sie Aufgaben planen über Systemsteuerung -> System und Sicherheit -> Verwaltung auf.
|
|
|
Erstellen Sie über Aktion -> Einfache Aufgabe erstellen... eine neue Aufgabe.
|
|
|
Vergeben Sie einen Namen und eine Beschreibung für die neue Aufgabe.
|
|
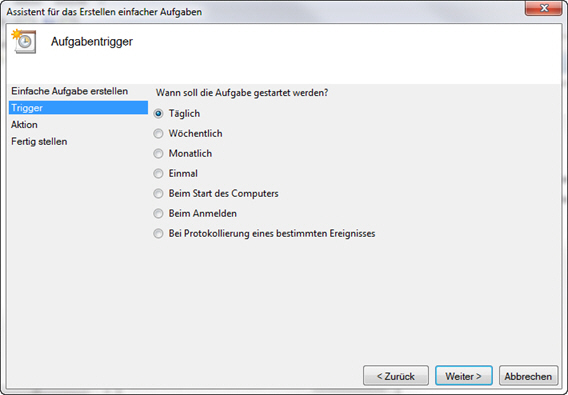
|
Dann legen Sie fest, wann die Aufgabe gestartet werden soll. In diesem Beispiel täglich.
Wenn Sie das R-Programm mehrmals täglich ausgeführt wollen, legen Sie einfach mehrere Aufgaben an!
|
|
|
Legen Sie das Datum für die erste Ausführung fest und die Uhrzeit für die wiederkehrende Ausführung.
|
|
|
Legen Sie fest, dass ein Programm ausgeführt werden soll.
|
|
|
Als auszuführendes Programm wird nicht die R-Skript/Program m-Datei eingetragen, sondern cmd.exe!
Die Batchdatei wird als Argument angegeben!
|
|
Das Argument:
|
Hier ein beispielhaftes Argument!
|
|
Nach Weiter wird Ihnen eine Zusammenfassung angezeigt und hier betätigen Sie die Fertig stellen-Schaltfläche!
|
Fertigstellen
|
|
Theoretisch sind Sie fertig, Sie müssen allerdings noch die Berechtigungen zur Ausführung des Programms einstellen!
Dazu markieren Sie in der Übersicht Ihr R-Skript und betätigen die rechte Maustaste und rufen die Eigenschaften auf:
|
Berechtigungen einstellen!
|
|
|
Wählen Sie “Unabhängig von der ...” aus und nach OK werden Sie nach Ihrem Passwort gefragt.
Nun sind Sie fertig und können die Ausführung testen!
|
|
Markieren Sie Ihr R-Skript in der Aufgabenübersicht, drücken die rechte Maustaste und wählen den Menüpunkt Ausführen. Die Skript-Ausführung werden Sie nicht wahrnehmen, aber das Resultat wird wie im Beispiel R im Batch-Modus im entsprechenden Verzeichnis abgelegt:
|
Testen!
|
|
Fertig! Das R-Programm wird auf Basis der von Ihnen festgelegten Parametern nun automatisch ausgeführt!
|

|
|
Rückgabe, Funktion
|
Gewöhnlich wird das Funktionsresultat über return(x) an die Konsole zurückgegeben. Diese Rückgabe ist natürlich keine Verpflichtung, wie schon in einigen Funktionen gezeigt. Als Zwischenlösung kann die Funktionsrückgabe über invisible(x) betrachtet werden.
D. h., dass komplette Funktionsergebnis kann an ein Objekt (z. B. a) übergeben werden:
a <- funktion(x)
Das Objekt a beinhaltet, wie gewöhnlich, das Funktionsresultat. Wenn Sie nur funktion(x) aufrufen, erhalten Sie keine Ausgabe! Sie wird durch invisible(x) unterdrückt.
|

|
|
Verzeichnis anzeigen
|
R bietet Ihnen Funktion zum Einlesen eines Verzeichnisses z. B. über die Funktionen dir() oder list.files(). Über das Attribut pattern kann der auszulesende Dateityp festgelegt werden. Möchten Sie nun Dateien vom Typ “csv” auslesen, gehen Sie sicher wie folgt vor:
Die Datei 7 (gelbe Markierung) ist allerdings nicht vom Typ “csv” sondern trägt csv in der Dateibezeichung. Um auch diese Dateien auszuschließen, müssen das Argument pattern um “\\” ergänzen:
Die Datei “lindacsv.lda.R” wird ausgeschlossen.
|

|
|
Zeit
|
Berechnungen mit der Zeit
Die Systemzeit, einschließlich Ihrer Zeitzone, wird über die Funktion
ausgegeben (CET = Central European Time). Über die Funktion ISOdate() und dem Parameter tz zur Einstellung der Zeitzone (tz=”” -> Zeitzone Ihres Computers), belegen Sie Variablen mit den von Ihnen gewünschten Zeiten:
> Zeit_1 <- ISOdate(2005,1,1, tz="")
> Zeit_1
[1] "2005-01-01 12:00:00 CET"
> Zeit_2 <- ISOdate(2008,11,8, 18,10,05, tz="")
> Zeit_2
[1] "2008-11-08 18:10:05 CET"
Möchten Sie Zeitdifferenzen berechnen, bietet R Ihnen die Funktion difftime() an:
> difftime(Zeit_2, Zeit_1)
Time difference of 1407.257 days
> difftime(Zeit_2, Zeit_1, units="weeks")
Time difference of 201.0367 weeks
Möchten Sie eine Datenobjekt mit einer Sequenz von beispielsweise 10 Tage erzeugen, können Sie dies mit der Funktion seq(as.Date()) erreichen:
> Tage <- seq(as.Date("2008-11-1"), by = "days", length = 10)
> Tage
[1] "2008-11-01" "2008-11-02" "2008-11-03" "2008-11-04" "2008-11-05"
[6] "2008-11-06" "2008-11-07" "2008-11-08" "2008-11-09" "2008-11-10"
Angeboten werden Ihnen folgende Varianten:
# Variante 1:
> Tage <- seq(as.Date("2008-11-1"), to = as.Date("2008-11-8"), by = "days")
> Tage
[1] "2008-11-01" "2008-11-02" "2008-11-03" "2008-11-04" "2008-11-05"
[6] "2008-11-06" "2008-11-07" "2008-11-08"
# Variante 2, vom Starttag um jeweils eine Woche weitergezählt:
> WochenTage <- seq(as.Date("2008-11-2"), to = as.Date("2008-11-30"), by = "weeks")
> WochenTage
[1] "2008-11-02" "2008-11-09" "2008-11-16" "2008-11-23" "2008-11-30"
Als Parameter für den by-Parameter sind neben den bereits gezeigten Argumente days und weeks auch die Argumente months und years zulässig.
|

|
|